點選藍字
今天推送的文章是2021年1月31日發表在International Journal of Biological Macromolecules上的“Improving the thermostability of trehalose synthase from Thermomonospora curvataby covalent cyclization using peptide tags and investigation of the underlying molecular mechanism”,通訊作者是南京工業大學的江凌教授和朱麗英副教授。
海藻糖是一種非還原性雙糖,由兩個葡萄糖分子透過α,α-1,1-糖苷鍵連線而成。海藻糖的化學性質有助於在各種應激條件下保護生物大分子,因此被稱為“生命之糖”。由於其在食品、化妝品、農業、醫藥和其他領域的高應用價值,透過生物方法生產海藻糖已引起越來越多的關注。目前,有三種主要的酶法生產海藻糖,其中海藻糖合成酶(TreS,EC 5.4.99.16)提供了生產海藻糖的最簡單途徑。實施這一過程的關鍵是應用具有高轉化率和適應性的TreS突變體。然而,目前商用的TreS突變體可能不適合海藻糖的工業生產,因為它們在高溫下穩定性差。
耐熱酶在提高工業規模生物技術工藝的成本效益方面發揮著重要作用。尋找具有高耐熱性的酶的最常見策略是從耐熱生物中挖掘基因,然而,耐熱酶的篩選通常效率低下。隨著蛋白質工程的發展,酶的定向進化和理性設計在開發工業應用耐熱酶方面取得了重大成功。然而,這些策略仍然需要耗時的最佳化,有時結果可能是微不足道的,這限制了它們的廣泛應用。因此,非常需要開發一種更有效和簡化的方法來改善酶的熱穩定性。
先前已經證明,透過SpyTag/SpyCatcher和SnoopTag/SnoopCatcher等系統進行蛋白質環化是一種非常有效的策略,可以產生耐熱酶。這些Tag/Catcher系統透過兩個短的多肽標籤導致蛋白質主鏈的特異性共價結合。目前已有多種酶與SpyTag/SpyCatcher環化,由此產生的環化突變體更耐熱,如熒光素酶、木聚糖酶、β-內醯胺酶、植酸酶等,這表明該技術在生物催化中具有巨大應用潛力,但是使用不同肽標籤環化的酶的穩定性增強機制和應用尚未詳細研究。
本研究中所選的T.curvata TreS代表了一種新型高效催化劑,在底物麥芽糖濃度高達30 g/L的情況下,其轉化率約為80%。然而,整個生物催化過程在30度下進行,在60度下熱處理30分鐘後,其一半活性喪失。在先前的研究中,位於活性位點附近的突變被證明是有效的。將T.curvata TreS序列與實驗室利用BLASTP建立的資料庫中的序列進行比對,最終從各種嗜熱細菌中篩選出5種不同的嗜熱TreS同源序列。作者使用UniProt分析了結果(https://www.uniprot.org),並鑑定了16個非保守氨基酸位點,包括Tyr21、His25、Phe28、Leu32、Gly78、Leu101、Asp115、Thr162、Phe182、Ala224、Ala265、Val289、Gly322、Ile323、Lys355和Cys506。突變位點與活性位點之間的距離在提高酶的熱穩定性方面起著關鍵作用。據報道,當距離小於10Å時,突變將導致酶活性的大幅波動。然而,在大多數情況下,當距離超過20Å時,未發現明顯的耐熱突變體。作者發現4個氨基酸殘基(即Gly78、Val289、Gly322和Ile323)與活性位點之間的距離為9-12Å。值得注意的是,I323L突變體的催化活性顯著提高,為了解釋該現象,使用SWISS-MODEL對其進行了建模(https://swissmodel.expasy.org),以5H2T(PDB ID)為模板。使用PyMol繪製野生型TreS和I323L的3D結構,並測量底物通道。野生型TreS的通道直徑為7.3Å,突變體為8.5Å。然而,底物麥芽糖的直徑為8.3Å,當底物進入催化中心時,在I323L突變後面臨較小的空間位阻,這可能是酶活性顯著提高的一個重要原因。與Gly78對應的嗜熱TreS同源蛋白序列中的氨基酸為天冬氨酸,與Val289和Ile323對應的氨基酸為亮氨酸,與GLY322對應的氨基酸為丙氨酸。因此,選擇G78D、V289L、G322A和I323L四種突變體進一步評估其熱穩定性。

之後對野生型TreS及其突變體進行鎳柱純化,SDS-PAGE結果表明突變不會影響酶的分子量,與野生型的分子量相同,約69kDa。相比之下,SpyTag-TreS-SpyCatcher、SpyTag-TreS-KTag和SnoopTag-TreS-SnoopCatcher、SnoopTagJR-TreS-DogTag的分子量分別為~89、~76、~87和~78 kDa。

半衰期(t1/2)是指酶活性降低50%所需的時間,是研究酶動力學穩定性的重要指標。野生型TreS在55°C下的t1/2僅為12.1分鐘,而環化TreS的t1/2比線性TreS高2到3倍。T50是酶活性降低50%的溫度,是研究酶熱穩定性的另一個重要指標。野生型TreS的T50為53.1℃,而環化TreS的T50值分別提高了7.5-15.5°C。與環化相比,G78D、V289L、G322A和I323L的t1/2值相對較低,分別為16.2、15.0、14.4和15.2分鐘,T50值分別為55.2、56.1、58.5和57.0℃。在pH值為7.2–7.4和40°C條件下,以麥芽糖為底物測量TreS突變體的動力學引數。環化TreS與天然線性TreS的Km值和Vmax值相似,表明催化效率不受環化影響。相比之下,G78D、V289L、G322A和I323L突變體的Km值不同程度增大,然而,突變體的Vmax值比野生型TreS高出2倍以上,表明突變體的酶活得到了提高。定點突變體和環化TreS的t1/2值和T50值的差異對應的p值小於0.01,表明突變和環化後熱穩定性顯著改善。基於這些資料,定點突變可以在一定程度上改善TreS的熱穩定性。然而,與四個環化TreS突變體相比,定點突變的改善效果稍差。主要原因可能是氨基酸在正確空間位置的定點突變機制可以區域性增加氫鍵和鹽橋的數量,從而在一定程度上幫助酶在高溫下保持其活性。然而,在更高的溫度下,肽標籤比定點突變對TreS具有更好的保護作用。因此,選擇環化TreS進行進一步分析。
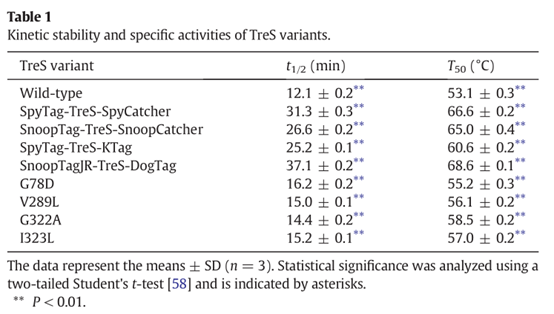
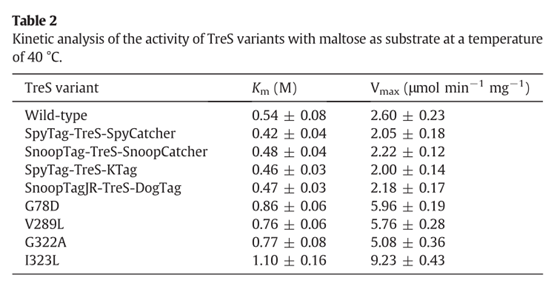
為了獲得環化TreS的結構,從PDB下載了天然TreS(PDB ID:5H2T)的晶體結構。用I-TASSER修復肽標籤SpyTag/SpyCatcher(PDB ID:4MLI),並從Streptococcus pneumoniae的RrgA晶體結構(PDB ID:2WW8)中分離SnoopTag/SnoopCatcher的結構模型。SpyTag/KTag和SnoopTagJR/DogTag採用相同的方法構建。PyMol用於獲得單殘基突變體的結構。作者利用編寫的程式碼,透過矩陣變換和旋轉,建立了SpyTag-TreS-SpyCatcher的結構模型。同樣的方法也用於獲得SpyTag-TreS-KTag、SnoopTag-TreS-SnoopCatcher和SnoopTagJR-TreS-DogTag的結構模型。
為了探索導致環化TreS熱穩定性增加的機制,在300和343 K的模擬溫度下進行了MD模擬。在300 K的模擬系統中,野生型TreS在15 ns後達到平衡,而環化TreS在超過25 ns後達到平衡。野生型TreS、SpyTag-TreS-SpyCatcher、SnoopTag-TreS-SnoopCatcher、SpyTag-TreS-KTag和SnoopTagJR-TreS-DogTag的RMSD值分別約為0.42、0.7、0.5、0.5和0.8 nm。當模擬系統的溫度增加到343 K時,野生型TreS的RMSD值逐漸增加,表明其結構變得不穩定。相比之下,SpyTag-TreS-SpyCatcher、SnoopTag-TreS-SnoopCatcher、SpyTag-TreS-KTag和SnoopTagJR-TreS-DogTag的RMSD值分別約為0.8、0.55、0.55和0.62 nm。環化TreS在模擬溫度300 K和343 K下的RMSD值相似,這表明環化TreS在不同溫度下是穩定的。Rg值分析結果則表明野生型TreS的三級結構在高溫下已被破壞,而環化TreS保持穩定,其結構變得緊湊,允許它們保持一定的活性水平。平衡的肽標籤和TreS將產生相對較強的相互作用力,這可以在高溫下保持TreS的三級結構,並防止疏水中心暴露在溶液中。
文章資訊:
PMID: 33285196
DOI: 10.1016/j.ijbiomac.2020.11.195
文章連結:
https://www.sciencedirect.com/science/article/pii/S0141813020351333?via%3Dihub





















