近日,中國農業科學院北京畜牧獸醫研究所奶產品質量與風險評估科技創新團隊成功建立了一種分析瘤胃微生物脲酶的靶向宏蛋白質組方法,為進一步揭示瘤胃脲酶蛋白多樣性和發展完善靶向宏蛋白質組學方法提供了指導。相關研究成果發表在《微生物前沿(Frontiers in Microbiology)》上。
宏蛋白質組學方法是研究微生物群落功能的重要技術方法,對於連線上游宏基因組和下游宏代謝組具有重要意義。靶向宏蛋白質組方法是指標對某一類功能微生物的某個或多個蛋白或酶,透過宏蛋白質組學方法體系,研究某一類功能微生物的多樣性與功能調控的方法,具有功能靶向性強、表型關聯性高和調控機制明顯等優勢。但是,目前靶向宏蛋白質組方法應用的較少,尤其是在瘤胃微生物功能研究方面鮮有報道。脲酶是瘤胃微生物生態系統中重要的酶,在尿素氮迴圈利用和微生物蛋白質合成中扮演重要角色,但是目前仍缺乏蛋白質水平的脲酶多樣性研究。
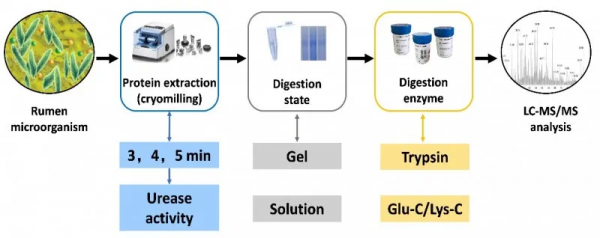
為了構建瘤胃微生物脲酶的靶向宏蛋白質組方法,研究人員透過檢測脲酶活性,優化了奶牛瘤胃微生物蛋白質提取(冷凍珠磨時間)條件,分析了瘤胃微生物高效酶解方法(膠條、溶液),優化了適合脲酶蛋白的消化酶種類(胰蛋白酶、蛋白內切酶穀氨酸-C、蛋白內切酶賴氨酸-C)。結果表明,液氮冷凍珠磨4分鐘時可提取到最高脲酶活性的蛋白質,膠內結合胰蛋白酶酶解方式能鑑定到最多的脲酶肽段。利用已知脲酶蛋白資料庫進行鑑定分析,發現瘤胃中存在大量新的未被認識的脲酶蛋白,並且脲酶蛋白多樣性較高。在鑑定的脲酶蛋白中,發現他們主要來自於原綠球菌和螺桿菌等。
該研究得到國家重點研發計劃、中國農業科學院重大任務和現代農業產業技術體系等專案資助。張曉音為文章第一作者,趙聖國和王加啟為文章通訊作者。
原文連結:https://doi.org/10.3389/fmicb.2020.573414
(來源:中國農業科學院北京畜牧獸醫研究所)