“有意思的是,我們把論文放到 bioRxiv 上以後,DeepMind 就把他們的蛋白複合物相關論文,也放到了 bioRxiv 上。”RoseTTAFold 最新 Science 論文的通訊作者叢倩表示[1]。
日前,兩大目前最先進的蛋白質預測工具 RoseTTAFold 和 AlphaFold,實現“隔空”聯合。
11 月 11 日,華盛頓大學蛋白設計研究所教授大衛·貝克(DavidBaker)團隊和德克薩斯大學西南醫學中心助理教授叢倩團隊聯合發表了最新 Science 論文《核心真核蛋白質複合物的計算結構》(Computedstructures of core eukaryotic protein complexes)。
論文共同通訊作者叢倩表示:“實際上該研究並沒有 DeepMind 的參與,但我覺得這種開放的競爭促進了領域發展。”
據悉,論文主要作者均來自貝克團隊,並由貝克和叢倩擔任通訊作者,後者是北大校友、此前是貝克實驗室的博士後,目前她已加入美國得克薩斯大學西南醫學中心工作,但仍和前者保持密切合作。
蛋白質和蛋白質的相互作用,在生物學中起著至關重要的作用,但許多真核蛋白質複合物的結構是未知的,並且可能有許多相互作用尚未確定。和谷歌旗下實驗室 DeepMind 的 AlphaFold 相對應的是,貝克團隊也提出了 RoseTTAFold(下稱 RTT)。
她和團隊在論文中寫道:“從預測單體到預測複合體,這使人類對蛋白質結構的理解邁出重要一步......我們的結果預示著結構生物學的新時代,在這個時代,計算在相互作用發現和結構確定中都發揮著重要作用。”
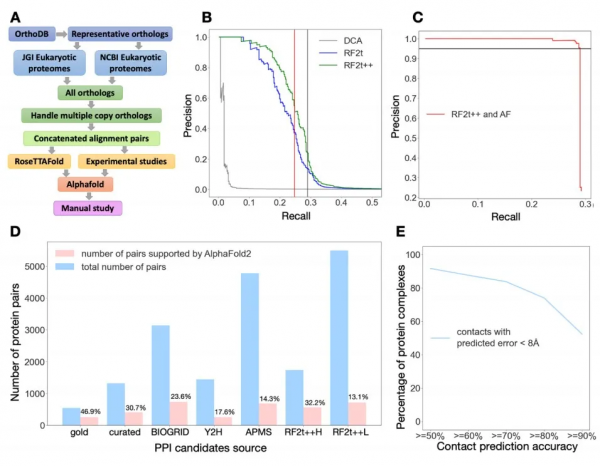
圖 | 蛋白質相互作用的評價和結構預測的準確性(來源:Science)
構建 700 多個相互作用蛋白的已知三維結構
透過結合 RTT 和 AlphaFold,研究人員得以預測出真核生物蛋白質複合體的三維結構。
以人類為例在我們體內,蛋白質單體們的相互作用至關重要。相互作用的蛋白形成複合體,有了它我們才得以完成各種生命運動。
但是,許多蛋白質複合體的結構都很不穩定,因此我們至今難以瞭解多數蛋白質複合體的三維結構。而此次成果,則讓科學家看到一線希望。
之前的研究中,叢倩主要藉助不同蛋白質的協同進化,去預測蛋白質組中可能相互作用的蛋白,雖然在原核生物中的準確性強於大規模試驗,但也需要大量序列,這意味著在已知序列較少的真核生物中難以得到應用。

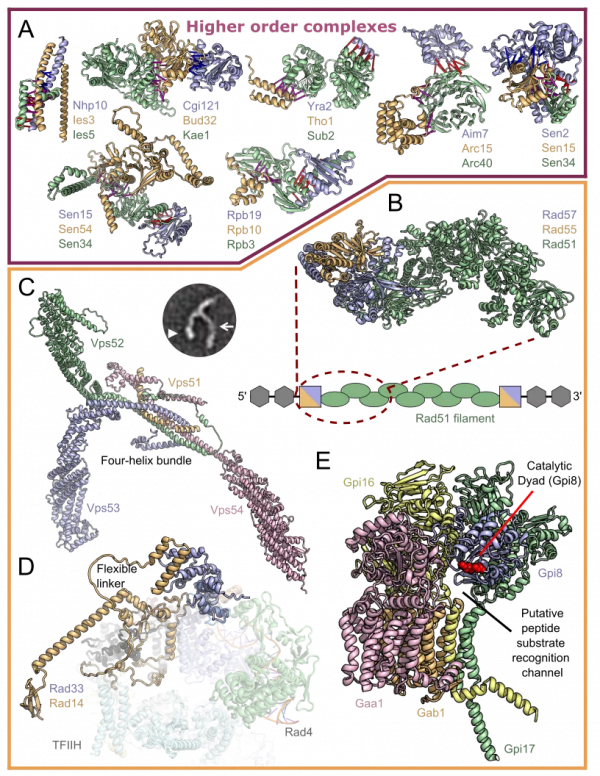
圖 | 參與轉錄、翻譯和 DNA 修復的蛋白質複合物(來源:Science)
和 AlphaFold 一樣,RTT 也能根據產生蛋白質的基因序列,來對蛋白質結構進行準確預測。不同之處在於。RTT 在 AlphaFold 的雙軌神經網路上,增添了第三個軌道。這讓 RTT 可同時兼顧蛋白質三維結構、氨基酸相互作用、和序列等資訊。
而在開發 RTT 時,貝克團隊使用了快速雙軌道模型,儘管準確率上還無法追平 AlphaFold 和三軌道模型,但勝在運算速度——大約是 AlphaFold 一百倍。
叢倩表示:“AlphaFold 開源了,所以我們拿來用了。”同時,她將兩者優勢進行結合,從而實現在全蛋白組的範圍內,篩選出存在相互作用可能性的蛋白質,並對其結構進行了預測。
據瞭解,雙軌 RTT 的速度相對更快,這和全蛋白質組的規模很契合。而且,RTT 更加依賴於蛋白質之間的共進化訊號,要知道能在細胞中相互作用的蛋白質,一定存在協同進化的特徵。相比之下,更側重於三維結構的 AlphaFold,也更適合去模擬兩個蛋白質在形成穩定複合體上的可能性。
此前的 RTT 和 AlphaFold,均用於預測蛋白質單體結構。將兩者進行拓展之後,即可預測蛋白質複合體的結構,還可在蛋白質組範圍內,去預測細胞內相互作用的潛在蛋白質。兩者的結合,也將預測相互作用的蛋白質、以及預測蛋白質複合體結構的準確性提升了一定幅度。以預測真核生物為例,準確率在 80%~90% 之間。
她選取了酵母菌作為該研究的研究物件,這是一種常見的真核模式生物。而且,其擁有較小的基因組、以及相對少見的 RNA 剪接,研究起來比較方便。此外,業內已積累大量酵母菌中蛋白質相互作用的資料,藉助這些資料可幫助核對預測結果準確性。
藉助不同蛋白質殘基的共同進化,叢倩從酵母菌裡篩查出 830 萬對蛋白質,藉此發現 1505 種 “疑似” 蛋白質複合體,而此前實驗已解析出其中 699 個蛋白質複合體的三維結構,而當前的工作預測了其餘 806 個蛋白質複合體的三維結構,包括 106 個首次被描述的全新蛋白質複合體。
據悉,儘管當前論文中報道的多數的三維結構模型只包含一對相互作用的蛋白,但是團隊也嘗試了給更大型的蛋白復,包含 3 到 5 個蛋白的複合體建模。這些複合體在真核細胞的多數關鍵過程中都發揮著作用,藉此可為生物學功能提供廣泛的見解。
此次 RTT 和 AlphaFold 的結合,也讓演算法實用性得以驗證。
據介紹,該論文對很多蛋白質複合物進行了深入研究,為了解細胞的功能提供了豐富的見解。例如,一種複合物含有蛋白質 RAD51,已知該蛋白質在人類 DNA 修復和癌症進展中起著關鍵作用。另一個包括知之甚少的糖基磷脂醯肌醇轉醯胺酶,它與人類的神經發育障礙和癌症有關。瞭解這些蛋白質和其他蛋白質如何相互作用可能為開發用於治療各種健康疾病的新藥物開啟大門。
透過進一步研究,叢倩還找到了具有諸多功能相關性的蛋白質複合體,這些複合體會參與蛋白質跨膜運輸、染色體分離、DNA 修復與轉錄翻譯、DNA 同源重組等生命活動。部分功能已知的蛋白質、和功能未知的蛋白質的相互作用也被發現。
叢倩表示,此次 RTT 的新成果、和 AlphaFold 的單體蛋白結構預測成果,彰顯了 AI 計算的重要性,也讓結構生物學進入了新時代。
她還認為,此次研究得以實現大規模預測生物體內複合體的結構,並能在蛋白質組、或者相互作用組的水平上,找出哪些蛋白應該會相互作用。這種研究思路區別於傳統的已知蛋白質相互作用、再去研究功能,而是已知生物功能、再去預測其他的可能在此功能通路上起重要作用的蛋白。
貝克也在其實驗室的新聞稿中表示:“隨著計算機方法變得越來越強大,生成大量科學資料比以往任何時候都容易,但要理解它仍然需要科學專家,所以我們招募了一群專家生物學家來解釋我們的 3D 蛋白質模型。這是最好的社群科學。”
未來將探索用於人類蛋白
“現在兩種工具都拓展到了蛋白複合物的預測,我們的重點是預測哪些蛋白會相互作用,DeepMind的重點是預測這些相互作用的蛋白會形成怎樣的三維結構,”叢倩說,“這項工作表明,深度學習也可對生物學中數十年的老問題產生真正的見解——不僅是特定蛋白質的樣子,還有哪些蛋白質聚集在一起相互作用。”她表示,該方法擴充套件了基於大規模深度學習的結構建模的範圍,從單體蛋白質到蛋白質組裝。
正如上述例子所強調的,跟蹤這裡提出的許多新的複合物將促進對真核細胞過程的廣泛理解,併為治療干預提供新的靶點。方法可以直接擴充套件到人類蛋白質組,但需要更多的計算時間,以及進一步提高方法的準確性,因為很多高等生物特有的蛋白之間的共進化訊號可能比較弱。
儘管如此,該成果也給人類蛋白質複合體研究、以及蛋白質組研究帶來了新希望。概括來說,該方法將基於大規模深度學習的結構建模範圍,從單體蛋白質擴充套件到了蛋白質組裝,對許多新複合物的後續研究應該會促進對廣泛的真核細胞過程的理解。
相信 RTT 和 AlphaFold 這兩支團隊都會不斷最佳化演算法,從而為識別人類細胞中未知的蛋白質複合物、並預測出相關結構進行努力,屆時很多疾病也有望迎來新療法。
另據悉,在這項工作中生成的蛋白質結構可從蛋白質資料庫模型檔案(ModelArchive)下載。叢倩和團隊也在論文中致謝了國際蛋白質結構資料庫(Protein Data Bank,PDB)的 John Westbrook(約翰·威斯布魯克)在建立格式和軟體程式碼以便將模型有效儲存到資料檔案中的支援,在準備這份手稿的過程中,約翰遺憾去世。
-End-
參考:
1、I. R. Humphreys et al., Science 10.1126/science.abm4805