隨著純生信的發展,幾年前的套路已經很難發表文章了,就像下面的這種套路文章:
The Cancer Genome Atlas (TCGA) based m6A methylation-related genes predict prognosis in hepatocellular carcinoma
分析內容操作步驟:
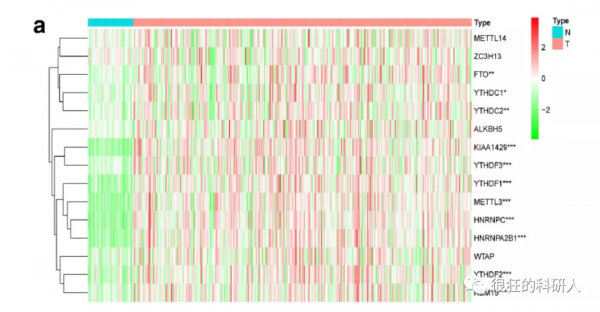
1、下載TCGA資料,提取出15個m6A基因基因矩陣,做差異分析,並且繪製熱圖
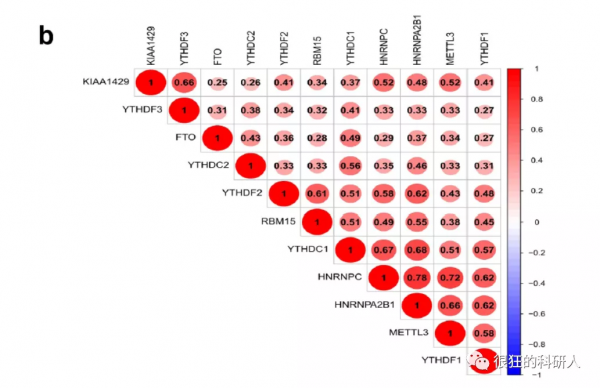
2、做相關性分析
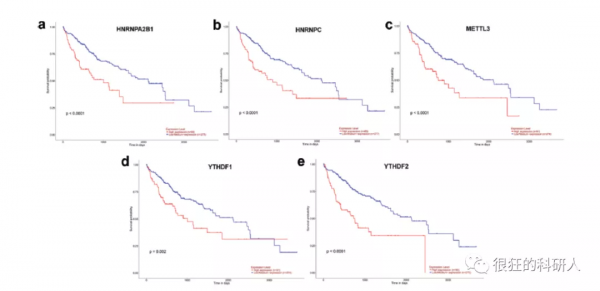
3、對差異的m6A基因做生存分析
4、進行腫瘤分型,根據一致性聚類與主成分分析的分型結果進行生存分析
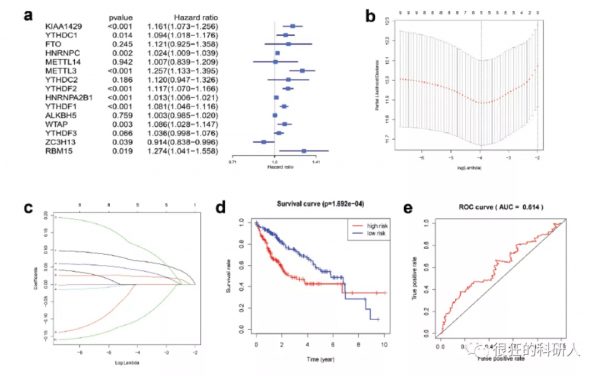
5、對15個m6A做單因素Cox迴歸,然後選出有意義的進行lasso迴歸進行進一步篩選,然後選到5個基因構建多因素Cox迴歸,計算risk score,根據高低risk score 繪製生存曲線和ROC曲線
5、將5基因模型的risk score聯合臨床進行單因素和多因素的Cox迴歸,並且繪製相關的熱圖
像上面這種文章在PubMed上可以檢索到一大堆,說明m6A這個熱點早已經爛大街了,現在很多期刊對這樣的文章都是比較反感的,因為覺得沒有什麼創新,因為別人把能做的都做了。如果你將基因替換一下,情況就不一樣了,你可以替換成其他甲基化修飾,例如m7G,這個熱點的純生信都是空白的,就等著大家去搶發這個熱點。
現在都是很卷的,大家都懂得了創新,就看誰的熱點最新、誰的速度快,誰就能佔得搶先發表的機會。現在連很多低分的期刊都需要看你的純生信文章是否創新,一看是比較舊的稿子,立刻秒拒,有不少做純生信的人都有這樣的體會。