jing 生信人
空間轉錄組(ST)能夠在單細胞或亞細胞水平上全面刻畫組織結構,幫助理解傳統技術無法獲得的生物學機制。過去的幾年已經開發出了各種各樣空間轉錄組分析技術,這些技術被Nature Methods評為2020年的最佳方法。因此,今天小編和大家分享一篇今年10月發表在Genome Research(IF:9.043)雜誌上的綜述。文章總結了空間轉錄組資料分析方法的現狀,並討論了它們如何在不同的技術平臺上執行,可以說一文帶你瞭解空間轉錄組,感興趣的小夥伴get起來吧。
Advances in spatial transcriptomic data analysis
空間轉錄組資料分析進展
一.文章背景
多細胞生物由組織和器官組成,組織和器官都專門從事一個生物過程。雖然正常細胞都擁有相同基因組,但它們的基因表達模式和形態可能截然不同。這種差異不僅是由內部基因調控引起,也是由外部組織環境發出的訊號引起的。儘管幾十年來基因組研究積累了大量細胞型別特異性基因調控的資訊,但目前對細胞外部環境相互的理解仍然有限。不過隨著近年來技術的爆炸式增長,已經實現了細胞異質性和組織及器官空間組織的系統級刻畫。其中最值得注意的是單細胞RNA-seq技術(scRNA-seq)的快速發展,使得在一個器官中描繪和比較細胞的基因表達模式成為可能。scRNA-seq在發現新細胞型別方面發揮了關鍵作用,併為建立不同物種的綜合細胞圖譜奠定了基礎。然而,由於實驗過程透過機械和酶解步驟創造單細胞懸浮液,這不可避免地破壞了原始的組織結構。因此,僅從細胞成分重建組織結構是極其困難的。
二.空間轉錄組主要技術及資料
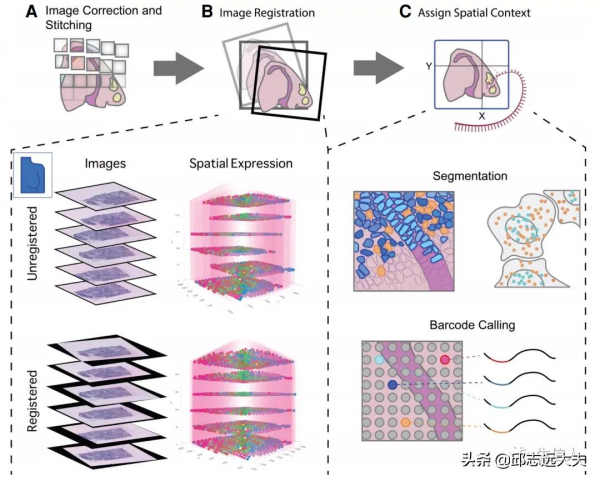
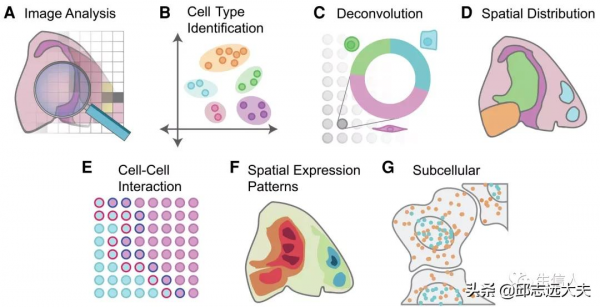
目前在單細胞解析度和全基因組規模上研究組織的方法主要有三種,分別為序列熒光原位雜交(FISH),及連線已知座標的所有轉錄本的空間標記和選擇基因用於原位測序(ISS)。文章主要使用了四個資料集(圖1)來說明不同空間轉錄組分析的結果或方法:1)乳腺腫瘤生物標本切片的全基因空間轉錄組資料集。2)全小鼠冠狀腦切片的亞細胞空間資料集。3)人類心臟全基因組空間資料集。4)老鼠體感皮層內數百個細胞的10000個基因的亞細胞空間資料。從原始ST資料集獲取基因表達矩陣和相應的空間座標不是一個簡單的過程,它包含許多預處理步驟。這些步驟通常依賴於技術或平臺,但有一些重複的步驟是固有的,例如基於成像資料的影象配準、拼接和細胞分割(圖2)。對於基於影象的ST資料,如FISH和ISS技術,最常見的影象處理步驟是影象校正、拼接、配準、分割,然後定位和解碼。另一方面,有些資料並不一定需要成像,而是透過在一個定義的空間單元內捕獲轉錄本,並在測序步驟之前將它們與一個已知的座標系統連線。因此,這些方法通常不依賴於上述影象處理步驟。然而,測序後還需要額外的步驟來將轉錄本映射回它們的空間座標。當有組織影象時,可以將其與空間座標系統疊加。不過無論技術如何差異,ST分析的一個共同目標是連線和整合來自基因表達和細胞轉錄本位置的資訊,這對於提取有用的生物資訊至關重要(圖3)。
圖1 文章使用的資料集
圖2 原始空間轉錄組資料的預處理
圖3 空間轉錄組學分析方法綜述
三.ST資料識別細胞型別
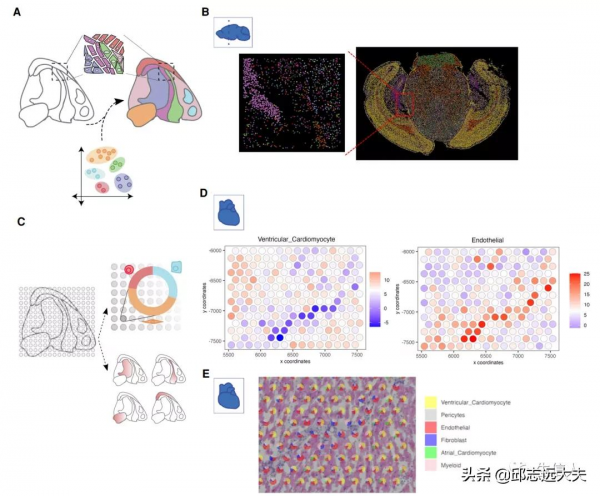
細胞型別的識別和定位是ST資料分析最基本的任務,因此在這一部分作者對ST資料識別細胞型別進行了介紹。如果資料有單細胞解析度,無監督聚類結合手動或自動註釋是識別細胞型別的常見方法(圖4A)。由於細胞型別識別不需要空間資訊,因此與scRNA-seq分析相似,如基於community的方法,如Louvain和Leiden聚類。為了說明,作者使用MERFISH冠狀切片資料集並應用Leiden聚類,總共得到19個不同的類。然後對這些簇進行註釋並映射回空間座標(圖4B)。目前有兩種估算細胞型別組成的方法(圖4C)。第一種方法是評估每個位點上表達基因中細胞型別特異性標記的富集情況。這種方法速度很快,每次可以執行一種細胞型別。然而,結果是定性的,表明存在或不存在一種細胞型別。第二種方法是去卷積,目的是定量估計每個位置不同型別細胞的比例。已經開發了許多去卷積方法用於RNA-seq資料分析,原則上也可以應用於ST分析,不過ST資料具有某些不同的特性,如與每個位置關聯的細胞數量很少。因此,通常使用專為ST分析設計的方法。在這些方法中,RCTD使用了基因計數的線性迴歸模型,進一步納入了針對平臺特異性變異的隨機效應項,基因表達水平採用泊松分佈模型。Cell2location使用類似的方法,但基因表達使用負二項分佈模型,它還可以模擬特定平臺和特定位置的效果。SpatialDWLS使用兩個步驟來減少噪聲:第一步使用富集分析來識別細胞型別,第二步使用衰減加權最小二乘法量化每種細胞型別的相對比例。SPOTlight使用種子非負矩陣分解(NMF)迴歸,並使用細胞型別marker基因和非負最小二乘(NNLS)進行初始化,用於後續的去卷積。DSTG使用基於圖的卷積網路。DestVI使用變化推理方法去卷積。作者使用Visium心臟資料集和匹配的scRNA-seq資料進行細胞型別富集(圖4D)和空間去卷積(圖4E),對這些方法具體說明。研究細胞型別定位的一種補充方法是使用scRNA-seq資料作為起點,然後根據空間表達輪廓的相似性重構空間資訊。在ST技術爆炸之前,使用傳統方法只能獲得少數標誌性基因的空間資訊。利用這些有限的資訊開發了tomo-seq和Geo-seq技術,能從2D切片中獲得的基因表達譜重建3D模式。不過這些空間資訊不是直接從資料中測量的,因此仍然是推測性的。隨著ST技術在過去幾年的快速發展,現在可以直接測量空間資訊,並進一步與scRNA-seq資料整合。因此,新的方法以更平衡的方式整合scRNA-seq和ST資料。例如,使用了與平臺無關的相互最近鄰(MNN)方法來對齊這些資料型別,從而生成細胞位置對映。其中DEEPsc使用人工神經網路來預測空間位置;GLUER結合NMF、MNN演算法和深度神經網路對資料進行對齊;Tangram將scRNA-seq和ST資料集對齊,同時優化了scRNA-seq資料和空間資料中每個基因之間的空間相關性。NovaSparc和D-CE也類似,不過對齊可以是機率的,也可以是確定性的。在已知細胞總數的前提下,Tangram對齊的確定性模式也可以作為一種去卷積方法。
圖4 利用空間轉錄組資料進行細胞型別識別的策略
四.刻畫轉錄組譜的空間模式
ST分析的關鍵不僅在於刻畫細胞型別,還在於它們的空間組織。這對於研究組織結構和細胞間相互作用至關重要(圖5A,C,E)。成對富集分析可以用於識別可能相鄰的細胞型別對,為了說明如何研究空間網路模式和細胞鄰近,作者使用MERFISH冠狀切片資料,基於每個細胞的物理座標建立了一個細胞鄰近網路。細胞-細胞鄰近網路連線熱圖顯示不同細胞型別之間的連線性及所研究組織的空間拓撲結構(圖5B)。圖5D顯示了對單個生態位的詳細探索,將特定的細胞識別為源,然後描述它們與其他鄰近細胞型別的連線。spatialDE使用一個隨機效應模型,該模型包含兩個terms,分別對應於空間成分和非空間成分。空間變數可以指定為各種形式,如線性、週期或高斯過程。空間變異性的程度由這兩項所解釋的方差比來量化。SOMDE就使用類似的方法,但透過使用自組織對映變換壓縮空間資訊來提高計算效率;Trendsceek將空間格局模型作為一個標記點過程;SPARK透過廣義線性空間模型對空間計數資料進行建模;有些方法主要考慮區域性連續性,如binSpect檢測空間相干基因,即傾向於在相鄰細胞中共同表達的基因。另一種方法是量化空間結構的擴散步驟。作者以MERFISH冠狀腦切片資料為例,利用binSpect識別具有空間相干模式的基因,排名靠前的基因如圖5F所示。
圖5 空間模式分析
五.亞細胞結構分析
在這一部分作者使用小鼠體感皮層資料集來說明亞細胞資料分析的一些關鍵概念(圖6)。在亞細胞解析度的資料集中,每個點通常代表一個單一的轉錄本(圖6A)。分析亞細胞基因表達模式可以作為空間分析的一種替代方法,也可以用於提高細胞分割的準確性(圖6B)。透過分析共定位模式(圖6C)和每個細胞內的轉錄動力學(圖6D),可以發現基因之間或基因與亞細胞結構之間的空間關係。許多方法嘗試利用亞細胞基因表達模式來規避細胞分割。如SSAM直接將細胞型別標籤分配給畫素,而不進行細胞分割。stLearn使用類似的方法,但將空間近端畫素進一步叢集。Spage2vec也使用了類似的方法,但採用了一種神經網路公式。另外,也開發了基於已知細胞型別特定簽名的監督細胞型別對映策略。如用樸素貝葉斯模型為HDST資料分配細胞型別。亞細胞基因表達模式可以反過來用於改善細胞分割。例如,Baysor模型利用馬爾可夫隨機場模型對亞細胞基因表達模式進行建模,並進一步整合細胞形狀標記資訊來提高細胞分割的準確性。Sparcle使用Dirichlet過程混合模型以及相鄰細胞與相鄰轉錄本之間的轉錄本距離來增強細胞分割。JTSA使用EM演算法迭代改進畫素及基因表達譜分類和細胞邊界標註。分析基因表達的亞細胞模式也可以提供新的生物學見解。如已經開發了一種原位RNA速度方法,利用亞細胞RNA定位資訊來推斷轉錄速率。由於新轉錄的RNA在細胞核中積累,而成熟的mRNA需要被運輸到細胞質中進行翻譯(圖6D),每個基因相關的核轉錄本與細胞質轉錄本的相對組成可以用來估計轉錄活性。此外,利用過氧化物酶APEX2對RNA進行直接鄰近標記(APEX-seq),可以高解析度地識別細胞質中共定位的mRNA種類。對結果資料的分析發現,共定位的RNA與已知的蛋白質共定位模式之間存在顯著的對應關係,這表明RNA共定位可能有助於區域性蛋白質翻譯和複雜的形成(圖6c)。
圖6 亞細胞解析度空間轉錄組學分析示意圖
六.細胞與組織環境溝通
ST分析的一個重要目標是研究細胞如何與組織環境溝通(圖7)。細胞行為可以透過直接的物理相互作用、分泌分子或與細胞外基質相互作用受到組織環境的影響(圖7A)。細胞間的通訊通常是空間協調的,並且具有高度的細胞型別特異性。因此,即使在相同的細胞型別中,細胞型別組成的變化也可能導致基因表達的顯著變化(圖7B,C)。R包Giotto採用雙向比較,比較同一細胞型別被不同相鄰細胞包圍的基因表達模式,來識別相互作用改變基因。與單獨使用基因表達資訊相比,使用空間資訊可以顯著減少假陽性配體受體活性預測的數量,CellPhoneDB中使用了類似的方法。在這一部分作者使用Cell2location來推斷不同細胞型別的位置,然後比較不同細胞鄰區相關的基因表達模式。也有其他方法用於量化相鄰細胞型別的影響,包括卷積神經網路。及將基因表達譜分解為空間分量和非空間分量,然後利用鄰域內的細胞型別組成來估計空間分量的方法。此外,也有演算法根據細胞與細胞的相互作用模式重建空間位置。
圖7 從配體-受體相互作用推斷細胞通訊
七.空間資料分析和視覺化工具
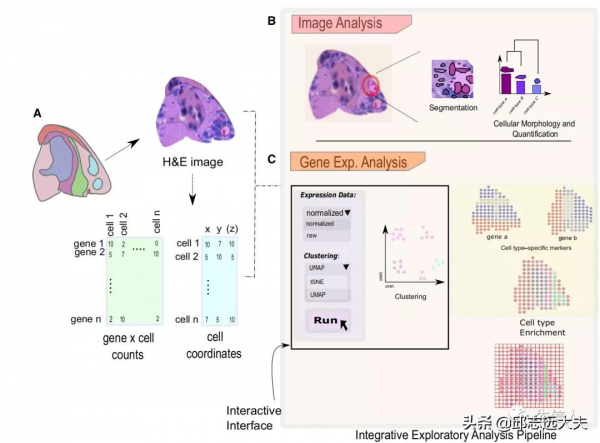
開發空間資料結構工具如今越來越重要,理想情況下匯入原始資料(圖8A)及影象分析(圖8B),和分析結果及資料準備發表(圖8C)都應該是在個人電腦上。這一部分作者介紹了下游資料分析工具。這些工具基本都是用R或Python編寫的。如R包Giotto可以用於各種各樣的ST技術,核心由一個專門為空間資料設計的物件組成。同時,Giotto提供了一個基於瀏覽器的視覺化工具,該工具允許使用者匯出他們獲得的結果,並互動式的探索空間資料集。Seurat作為一個流行scRNA-seq分析R包為人所熟知,它也開始提供一些針對空間資料視覺化和識別空間表達模式的功能。此外其他工具如STUtility和SPATA也建立在Seurat資料結構之上建立了更全面的管道,這些管道目前僅適用於ST技術。STUtility是專為ST技術開發的,提供了多種成像和資料分析方法。SPATA也關注ST資料,其開發目的是促進Seurat和Monocle整合。除了視覺化和常見的資料分析功能,SPATA還可以識別或描繪空間軌跡。Squidpy是空間版的SCANPY, SCANPY是用於scRNA-seq分析的Python庫,它還提供影象層面的分析。Stlearn是另一個用於ST資料分析的Python庫,它專注於將基因表達和影象資訊整合。這些包或工具大多數都是獨立的實驗室開發的,導致這些資料結構不一定共享相同的資料格式。因此R/Bioconductor團隊精心設計普遍適用的資料結構,並於最近釋出了spatialExperiment class的第一個版本。這是一種新的S4類,它擴充套件了singleCellExperexperiment class,能用於操作幾種型別的ST資料集。一些R包已經開始使用這種資料結構,如SpatialLIBD和Spaniel,它們都擅長建立互動式程式來視覺化ST資料集。總之,這些努力可以在未來促進不同工具之間的互動操作性。
圖8 互動式探索性分析管道概述
到這裡這篇文章的主要內容就介紹完了,文章對空間轉錄組資料分析方法進行了總結,並使用空間轉錄資料集進行了舉例說明。近幾年來空間轉錄組領域快速發展,是一個熱點方向,感興趣的小夥伴不要錯過呀。
參考文獻
1.Advances in spatial transcriptomic data analysis;
2.High-throughput spatial mapping of single-cell RNA-seq data to tissue of origin;