責編 | 王一
隨著地球生物基因組計劃 (Earth BioGenome Project) 針對上百萬個真核生物基因組測序的開展,將有越來越多的複雜基因組被測序、拼裝。基因組比對是對這些基因組的最核心分析之一。包括很多農作物在內的許多非模式生物的基因組比人、擬南芥等模式生物的基因組要複雜很多。針對哺乳類動物開發的比較基因組學工具在應用到複雜的基因組時往往不能產生符合生物學預期的結果。
1)很多親緣關係較近的農作物和野生物種之間存在全基因組複製和染色體重新排列組合等差異。“多對多”或者“一對一”的基因組比對策略不符合已有的全基因組複製研究結果。
2)基因組中很多保守的重要功能元件較短,比如轉錄因子結合位點的核心motif只有6-7bp。使用K-mer的啟發式演算法會丟失掉這部分序列。
3)使用啟發式演算法進行全基因組序列比對,多個複製的轉座子和其他機制形成的重複序列會引起很多假陽性的比對結果。
4)轉座子的跳躍和衰減會產生50bp到幾千bp長度的插入和缺失。常用的區域性序列比對策略不能跨越這部分較長的插入或缺失。
美國康奈爾大學Buckler實驗室和西班牙巴塞羅那超算中心聯合開發了針對這些複雜基因組比對問題的工具—— AnchorWave。AnchorWave使用蛋白編碼基因等保守序列作為錨定位點,設計了一個最長路徑演算法分析兩個基因組的共線性區域。使用目前最快的全域性序列比對演算法WFA設計了一個2-piece affine gap cost的比對策略,並透過WFA對每個錨定位點區和錨定位點間區進行單鹼基解析度的序列比對,進而獲得每個共線性區域的序列比對和全基因組水平的序列比對。
AnchorWave演算法流程圖
使用AnchorWave比對B73和Mo17兩個玉米基因組,可以把基因組的比對率從60-70%提高到99%。AnchorWave能將大約87%已報道的轉座子差異位點比對成完整的單個插入或缺失。而目前常用的基因組比對工具的轉座子差異位點召回率(recall)為0。
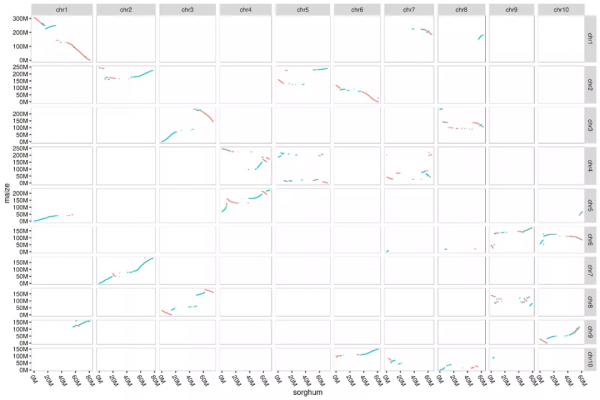
透過比對高粱和玉米基因組、大豆和其野生物種基因組、番茄和馬鈴薯基因組顯示AnchorWave能產生符合全基因複製進化預期的序列比對深度。顯著提高調控序列區域的比對率,並降低轉座子區域的假陽性。
和高粱基因組相比玉米基因組多一次全基因組複製,AnchorWave能產生比對深度為2:1的結果。
最後作者也使用人類和黑猩猩的基因組對AnchorWave進行了測試,AnchorWave產生的結果略優於目前應用最廣泛的net-chain流程。
綜上所述,AnchorWave在對以農作物基因組為代表的複雜基因組比對時顯著的優於目前已有的比較基因組學工具。
論文連結:
https://www.pnas.org/content/119/1/e2113075119
AnchorWave 軟體連結:
https://github.com/baoxingsong/anchorwave
本文來自BioArt植物微信公眾號,更多生物領域前沿資訊等你來發現!
轉載須知
【原創文章】BioArt植物原創文章,歡迎個人轉發分享,未經允許禁止轉載,所刊登的所有作品的著作權均為BioArt植物所擁有。BioArt植物保留所有法定權利,違者必究。