導 語
近年來,空間轉錄組(Spatially Resolved Transcriptoms, SRT)技術的進步使得利用組織中的空間資訊進行基因表達譜分析成為可能。目前,關於SRT的實驗策略大致可分為兩類:第一類是具有單細胞解析度的原位雜交或測序技術(包括seqFISH、MERFISH等),能夠檢測單個細胞中數百到數千個基因的表達水平;第二類是基於原位捕獲的技術(包括SLIDE-seq和10x Visium等),即先進行空間條形碼編碼再進行測序,可以檢測捕獲特定位置中數千個基因的表達水平。這些不同的SRT技術使得揭示異質組織的複雜轉錄結構成為可能,並增強了人們對疾病細胞機制的理解。
在SRT研究中,一個重要的步驟是區分不同的組織區域,即定義在基因表達和組織學上空間一致的區域。傳統的聚類方法,如K-means、Louvain等通常只能將基因表達資料作為輸入,由於缺乏對空間資訊和組織學的考慮,由此產生的聚類可能不具有整體性和連續性,不能反映真實的組織結構。
SRT研究中另一重要環節是鑑定出空間變異基因(Spatially Variable Genes, SVGs),將空間結構域與生物功能聯絡起來。已知的用於檢測SVG的方法,如Trendsceek、SpatialDE和SPARK等只能獨立地檢測每個基因,並返回一個P值來表示基因的空間特異性,同樣出於對空間結構考慮的缺乏,這些方法檢測到的基因並不能保證特定的空間表達模式,因此難以利用這些基因進行後續深入的生物學研究。
為了同時解決上述兩個難題,近日,美國賓夕法尼亞大學佩雷爾曼醫學院生物統計系李明瑤教授課題組在Nature Methods期刊線上發表了題為“SpaGCN: Integrating gene expression, spatial location and histology to identify spatial domains and spatially variable genes by graph convolutional network”的文章,提出了空間轉錄組資料分析的新演算法SpaGCN。SpaGCN首先構建表示資料空間依賴性的無向加權圖,再將基因表達、空間位置和組織學進行整合來識別空間域。此外,SpaGCN還可以檢測每個空間域豐富的SVG,透過將搜尋空間限制在空間域上,以保證檢測到的SVG都具有空間表達模式。SpaGCN適用於分析多種型別的SRT資料,包括ST、10x Visium、SLIDE-seqV2等。
文章發表在Nature Methods上
主要研究內容
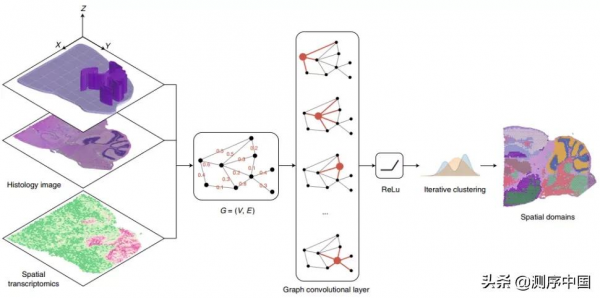
SpaGCN演算法工作原理
SpaGCN是一種利用圖卷積網路分析空間轉錄組資料、劃分不同組織區域並尋找區域富集基因的機器學習演算法。SpaGCN首先透過構建一張加權無向圖(Undirected Weighted Graph)將空間轉錄組中的基因表達和空間資訊進行整合;隨後利用一個圖卷積層來聚集來自相鄰點的基因表達資訊,並把資訊輸送到一個分類層將整個組織劃分成不同的區域;同時使用無監督迭代聚類演算法對這些鄰近點進行聚類並劃分為特定空間域。
圖1. SpaGCN演算法工作原理概圖,來源:Nature Methods
接下來,SpaGCN會在鑑定出來的空間域中尋找特異富集SVG,以確保找出的基因具有相似的表達模式,可用於進一步研究該組織區域的功能。當單個基因不能標記一個結構域的表達模式時,SpaGCN將構建一個由多個基因組合而成的複合基因來表示該結構域的表達模式。
圖2. SpaGCN演算法工作原理概圖,來源:Nature Methods
利用SpaGCN分析人類原發性胰腺癌資料
為了證明納入組織學資訊的重要性,研究團隊分析了使用ST技術生成的人類原發性胰腺癌資料,該資料集包括224個點和16,448個基因,以及三個手動註釋的組織區域。組織學影象顯示了癌區和非癌區之間的明顯差異,表明組織學對聚類有參考價值,而SpaGCN具有對組織學建模的靈活性引數,該引數能夠在檢測每個點的鄰域時賦予組織學的權重。透過預設值,SpaGCN即可很好地檢測到非癌區域;當將組織學權重增加時,SpaGCN不僅能檢測到非癌區域和癌區域,還能檢測到與手動註釋的癌症區域一致的區域。以上實驗結果充分表明,SpaGCN將組織學資訊納入聚類中是有用且必要的。
圖3. SpaGCN在檢測空間域層面的效能比較,來源:Nature Methods
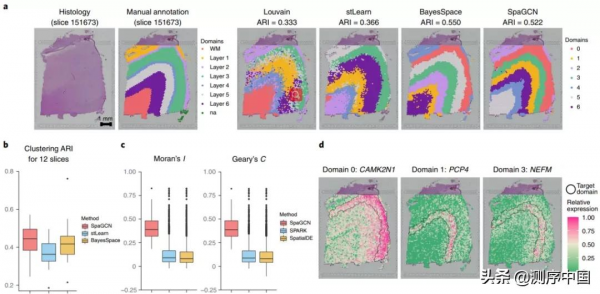
SpaGCN應用於人類背外側前額葉皮質資料
為了定量顯示SpaGCN在空間域檢測方面優於Louvain、stLearn和BayesSpace等演算法,研究團隊分析了使用10x Visium生成的人類背外側前額葉皮質資料,這項研究對三個人腦中的12個組織切片進行了測序,這些切片跨越六個神經元層和人類背外側前額葉皮質中的白質。結果顯示,SpaGCN和BayesSpace分析得到的空間域比Louvain更符合手動註釋的組織分割槽;stLearn利用了組織學資訊,但其效能與Louvain、SpaGCN和BayesSpace相比仍然較差。
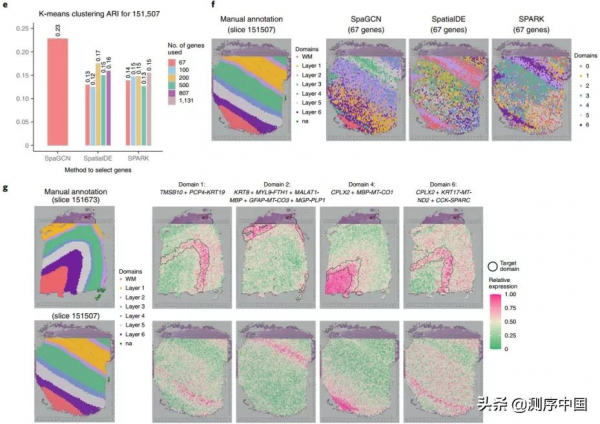
為了進一步驗證已識別的空間域,研究團隊計算了每個域的SVG。儘管SPARK和SpatialDE檢測到的SVG數量遠大於SpaGCN,但這兩種方法檢測到的基因無法區分不同程度的空間表達變異。SpaGCN檢測到的SVG的Moran's I值顯著高於SpatialDE和SPARK。
圖4. SpaGCN在檢測空間域層面的效能比較,來源:Nature Methods
接下來,研究團隊將SpaGCN、SpatialDE和SPARK檢測到的SVG進一步分析發現,與SpaGCN相比後兩者檢測到的基因缺乏空間模式,證明了SpaGCN檢測到的SVG的準確性和優勢。此外,在單個基因難以鑑定的時候,SpaGCN能夠找到特定領域的複合基因。
圖5. SpaGCN在鑑定SVG層面的效能比較,來源:Nature Methods
結 語
研究團隊推出了一種結合基因表達、空間定位和組織學的方法——SpaGCN,能夠來模擬基因表達的空間依賴性,並用於識別空間區域和鑑定空間富集SVG。效能驗證實驗的結果一致表明,SpaGCN可以識別具有一致基因表達和組織學的空間域,並且檢測到的SVG具有更清晰的空間表達模式和生物學功能相關性。隨著空間轉錄組在生物醫學研究中的重要性和日益普及,研究團隊希望SpaGCN的誕生能對研究人員的大規模空間轉錄組資料分析提供幫助。
參考文獻
1. Hu J, et al. SpaGCN: Integrating gene expression, spatial location and histology to identify spatial domains and spatially variable genes by graph convolutional network. Nat Methods. 2021 Nov;18(11):1342-1351.
2. Eng, C. L. et al. Transcriptome-scale super-resolved imaging in tissues by RNA seqFISH. Nature 568, 235–239 (2019).
3. Zhao, E. et al. Spatial transcriptomics at subspot resolution with BayesSpace. Nat. Biotechnol. https://doi.org/10.1038/s41587-021-00935-2 (2021).