導語
大家好,這次給大家介紹一篇2021年5月19日倫敦大學學院發表在BIB的一篇文章《Utilizing graph machine learning within drug discovery and development》本文的第一作者為Thomas Gaudelet 通訊作者為Jake P Taylor-King。
圖機器學習 (GML) 因其能夠模擬生物分子結構之間的功能關係以及整合多組學資料集(以及其他資料型別)而在製藥和生物技術行業中越來越受到關注。雖然該領域仍處於新興階段。但是, 研究表明,GML 將成為生物醫學機器學習中首選的建模框架。因此在這篇文章中,作者梳理了圖機器學習在生物醫藥領域的重要應用。
1介紹
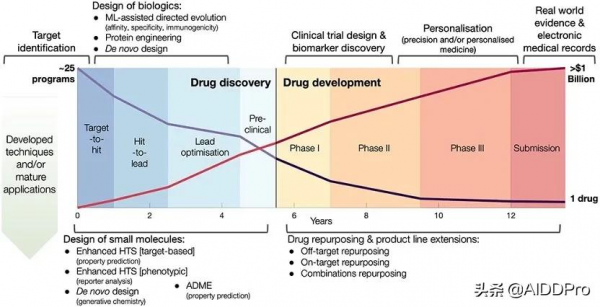
從藥物發現到上市的過程時間和資金消耗巨大因此人們對應用計算方法來加快藥物發現產生很大的興趣。
在藥物發現過程中,產生和使用的生物醫學資料的關鍵特徵之一是其相互關聯的性質。這種資料結構可以表示為圖形;這種數學抽象,廣泛用於生物學的各個學科和領域,以模擬在不同尺度上進行干預的生物實體之間的各種相互作用。在分子尺度上,蛋白質和其他生物分子可以表示為捕獲其氨基酸殘基之間的空間和結構關係的圖,而小分子藥物可以表示為其組成原子和化學鍵結構相關的圖。在中間尺度上,相互作用則是捕獲生物分子物種(例如代謝物、mRNA、蛋白質)之間特定型別相互作用的圖。其中蛋白質-蛋白質相互作用(PPI)圖則是最常見的一類。最後,在更高的抽象級別,知識圖譜可以代表藥物,副作用,診斷,相關治療和試驗結果之間的複雜關係。
GML方法的基本思想是學習節點(例如社交網路中的使用者)、邊(例如預測推薦系統中的未來互動)或整個圖(例如預測分子圖的屬性)的有效特徵表示)。在生物醫學領域,GML技術應用廣泛,透過知識圖挖掘圖結構資料,其包括藥物-靶標的相互作用和關係預測、分子特性預測、包括吸收、分佈、代謝和排洩 (ADME) 曲線的預測、到從頭分子設計。因此,GML方法在整個藥物開發的應用都中非常重要。
2圖上的機器學習
大多數對圖上的機器學習方法可以分解為兩個部分:通用編碼器和特定於任務的解碼器。編碼器將圖的節點或圖本身嵌入到低維特徵空間中,要嵌入整個圖,通常首先嵌入節點,然後應用置換不變的池化函式來生成圖級別表示(例如sum、mean、max)。解碼器計算相關任務的輸出。這些元件可以在兩步框架中組合使用,通常來說編碼器在無監督設定中進行預訓練,或者以端到端的方式進行。最終任務可以按照多種二分法進行分類:監督/無監督、歸納/轉導和節點級/圖級。這其中還可以將GML分為三類:
傳統方法:
圖統計(最短路徑,子結構)、隨機遊走(deepwalk、node2vec)透過遊走路徑作為句子透過跳詞模型來訓練出特徵向量。
幾何方法:
知識圖譜的嵌入矩陣/張量分解(對連線矩陣進行分解得到稠密的向量作為節點特徵)。
圖神經網路:
圖卷積、圖注意力、圖池化、關係圖卷積、訊息傳遞機制、關係圖卷積網路等。
3藥物開發應用
GML 如何應用於藥物發現和開發過程中的不同階段在下表中有詳細體現。
靶標識別
靶標識別是尋找在疾病的病理生理學中具有重要功能作用的分子靶標,以便假設藥物可以調節所述靶標,最終產生有益效果。早期的靶標包括 G 蛋白偶聯受體 (GPCR)、激酶和蛋白酶。
GML可以透過相互作用的生物分子的網路檢視,統一對疾病生物學、表型的遺傳驅動因素的現象學觀察。最終確定一個“可成藥”的干預點。
從系統生物學到圖上的機器學習
生物體或生物系統是由多個尺度的實體之間複雜且動態的相互作用組成。通常,系統生物學專注於具有靜態節點和邊緣的網路,而忽略了節點所代表的潛在生物分子的真實表徵。使用 GML,我們可以解釋包含多個相關尺度的更豐富的生物學表示,例如,分子結構的圖形表示、知識圖中的功能關係。此外,GML 可以從資料中學習圖形,而不是依賴於預先存在的不完整知識。隨著獲取生物資料成本的降低,GML的方法能幫助我們進一步認識生物系統的複雜性。
分子性質預測
製藥公司可能會針對特定目標篩選數百萬個小分子,然而,最終篩選的結果將透過藥物化學家進行結構最佳化,因此應該旨在透過僅篩選具有代表性的分子選擇來大幅減少搜尋空間,以便進行後續最佳化。一種方法是使用 GML方法選擇具有異質化學特性的分子。其中,使用藥物的圖形表示已經顯示出相對於沒有基於圖的分子特性預測任務方法的實質性改進。例如 QM9和MD17效能最好的方法都是基於 GNN。
從頭設計分子
相比之前討論的任務,更具挑戰性的任務是從頭開始小分子的從頭設計;也就是說,對於一個固定的目標(通常透過 3D 結構表示)設計一個合適的和具有選擇性的類藥物化合物實體。可以說肯定的說,尋找現有分子比從頭設計一個更簡單。
例如,已經可以透過使用變分自編碼器樣式架構來建立DDR1的新型化學分子。而透過GML技術和原有技術相融合能更好的輔助從頭設計藥物,例如透過蛋白質殘基的成對空間距離構建鄰接矩陣,這些方法構建的影象將用於後續的GML演算法框架來最佳化設計藥物的方法。
機器學習輔助的定向進化
GML 架構還沒有很多的應用於這個領域,但最近也有了一部分工作。例如,有課題組報道了在 70 000 種熒光素酶樣氧化還原酶的氨基酸序列上訓練了多個變分自動編碼器,並透過它生成了具有新功能的變體細菌熒光素酶。
蛋白質工程
最近,相關工作討論了使用基於 GNN 架構來預測蛋白質 - 蛋白質複合物的介面中涉及哪些氨基酸殘基的能力。每個蛋白質都被表示為一個圖,其中節點對應於氨基酸殘基,邊將每個殘基連線到它最接近的殘基。作者提出了具有不同複雜性的多個聚合函式,並遵循擴散卷積神經網路的一般原則。兩種蛋白質的每個殘基的輸出嵌入都被串聯起來。
從頭設計蛋白質
生物工程的一大目標是從頭開始設計蛋白質。使用基於圖的結構表示來利用自迴歸自注意力模型來預測相應的序列的工作也有相關的報道。
這一領域比較有名的模型為DeepFRI,這是一種基於結構和序列資訊預測蛋白質功能註釋的模型。模型作者根據蛋白質殘基之間的接觸圖定義了蛋白質的圖形表示。他們首先使用一個預訓練模組,然後將每個蛋白質的圖輸入到多個 GCN 層,最終輸出蛋白質的嵌入向量,透過MLP層,給出最終的功能預測。
藥物再利用
脫靶再利用
估計表明,每個小分子可能與數十個甚至數百個蛋白質相互作用。由於小分子的多效性,現有藥物的脫靶效應可能是尋找新適應症的一種有效方式。
現在有越來越多的 GML 文獻報道的方式可以在依賴或不依賴 3D 蛋白質結構的可用性的情況下推斷缺失的藥物-靶點相互作用。同時許多的用於藥物-靶標相互作用的 GML 方法不需要蛋白質結構。
討論
雖然GML在生物醫學的應用裡面前景廣闊,但是,GML 仍處於起步階段,許多的問題還亟待解決。例如,更深的 GNN 會過平滑效應和資訊過度壓縮的問題。最後,由於 GML 可以利用表示非結構化的多模態資料集,未來科學家一旦在資料整合方面取得巨大進步後,它或許能爆發出更大的能量。
參考文獻
Thomas Gaudelet, Ben Day, Arian R Jamasb, Jyothish Soman, Cristian Regep, Gertrude Liu, Jeremy B R Hayter, Richard Vickers, Charles Roberts, Jian Tang, David Roblin, Tom L Blundell, Michael M Bronstein, Jake P Taylor-King, Utilizing graph machine learning within drug discovery and development, Briefings in Bioinformatics, 2021;, bbab159, https://doi.org/10.1093/bib/bbab159
宣告
本文系AIDD Pro接收的外部投稿,文中所述觀點僅代表作者本人觀點,不代表AIDD Pro平臺,如您發現釋出內容有任何版權侵擾或者其他資訊的錯誤解讀,請及時聯絡AIDD Pro(請新增微訊號plgrace)進行刪改處理。