單核苷酸多型性(SNP)和插入/缺失(InDel)是人類基因組中最常見的兩種遺傳變異型別。在利用新一代高通量測序資料研究基因組變異和基因組功能時,SNP和InDel的檢測基本檢測專案。目前,已有多種不同演算法可用於短讀長測序資料中的SNP和InDel分析。但由於這些方法是針對短讀長測序資料開發的,因此在錯誤率高的長讀長測序資料上不能很好地執行。此外,短讀長測序固有的技術限制使得短讀長測序資料不能用於複雜或重複基因組區域的SNP和InDel檢測。
與短讀長測序技術相比,長讀長測序技術成本更低,reads長度更長,可以克服短讀長測序無法解決的多個挑戰性問題,已成功用於對不同物種的基因組進行測序。但與短讀長測序資料相比,長讀長測序的精準度較低,檢測錯誤率也更高。有研究表明,利用基於深度學習的演算法,在長讀長測序資料上可以精確檢測變異。根據單倍型資料的分階段比對可提高變異識別的準確性,現有的三種演算法(DeepVariant、Clairvoyante和Clair)在短讀長和長讀長資料上都能很好地執行單倍型資料比對,但這些演算法在SNP檢測中都忽略了來自遠端單倍型SNP的重要資訊。
近日,費城兒童醫院王凱教授團隊開發了一種新的深度學習演算法--NanoCaller,可充分利用長讀長測序在基因組區域中檢測變異,並在Genome Biology上發表了題為“NanoCaller for accurate detection of SNPs and indels in difficult-to-map regions from long-read sequencing by haplotype-aware deep neural networks”的研究文章。NanoCaller可利用單倍型資訊檢測SNP,使用稱為SNP的長reads進行定相,並透過區域性重新排列檢測InDel。研究團隊利用NanoCaller檢測了一個被廣泛使用的基準基因組中的41個全新變體,這是此前其他方法無法實現可靠檢測,有助於從長讀長測序中發現複雜基因組區域的新變體。
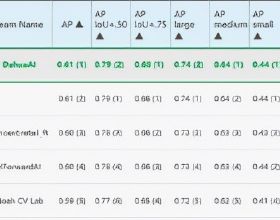
NanoCaller是將長讀長測序資料與參考基因組的比對作為輸入,根據指定的最小覆蓋率閾值和替代等位基因頻率閾值或插入/刪除頻率閾值選擇候選SNP或InDel位點(圖1)。
圖1. NanoCaller原理概述。來源:Genome Biology
為評估NanoCaller的效能,研究團隊將NanoCaller在牛津奈米孔測序資料中的變異檢測效能與Medaka、Clair 和 Longshot這三種現有演算法進行了比較。跨基因組分析對於證明變異檢測演算法的效能至關重要,將基於變異檢測演算法的機器學習模型在一組基因組上進行訓練,並在其他基因組進行測試。結果顯示,NanoCaller獲得的F1-score高於其他三個演算法,表明NanoCaller的效能優於其他演算法。(圖2a,b;F1-score經常被用來判斷演算法的精確度,能同時考慮精確率和檢測率)
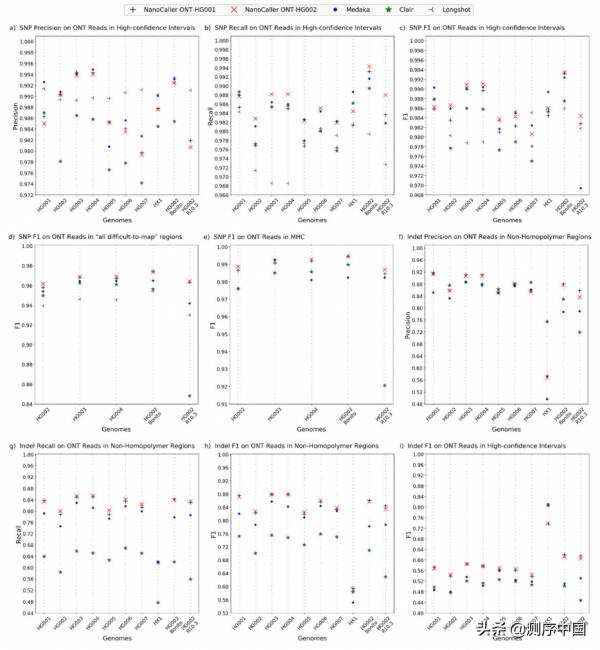
圖2. NanoCaller 和其他演算法在10個牛津奈米孔測序資料集中的效能。來源:Genome Biology
隨後,研究團隊將NanoCaller在PacBio測序reads資料上的變異檢測效能與三種現有演算法進行了比較。結果顯示,NanoCaller的表現(圖3)優於其他三個演算法,顯示出更強的競爭力。

圖3. NanoCaller和其他演算法在10個PacBio資料集上的效能。來源:Genome Biology
研究團隊還分析了NanoCaller在特定基因組上進行的SNP呼叫效能。透過分析Sanger測序結果,研究人員確定了41個全新變異(25個SNP、10個插入和6個缺失)。基於41個新變異,透過不同演算法進行了變異識別評估,以瞭解更準確的長讀長測序如何改善變異識別。結果顯示,NanoCaller能夠正確識別20個SNP,6個插入和2個缺失,其中10個SNP和1個缺失未被其他演算法正確檢測到。這可能是由於NanoCaller獨特的單倍型識別功能,以上結果證明了NanoCaller對SNP檢測的強大效能。

圖4. NanoCaller檢測到的新變異。來源:Genome Biology
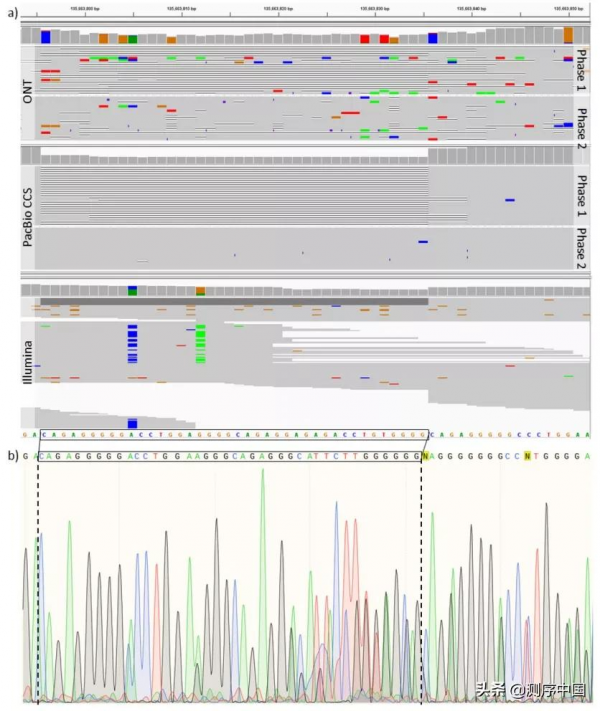
對於NanoCalle在InDel檢測方面的效能,研究發現NanoCalle可以檢測到無法被短讀長測序檢測到的變異。結果顯示,NanoCaller在chr9:135663805處檢測到缺失,其他演算法在chr9:135663799處檢測到缺失。如圖6a所示,與幾乎沒有證據支援缺失變異的短讀長測序資料相比,NanoCaller可提供缺失變異的準確資訊。
總之,該研究展示了一種深度學習演算法——NanoCaller,將單倍型結構整合到深度卷積神經網路中,用於長讀長測序資料檢測SNP,並使用多序列比對重新檢測插入缺失候選位點。與其他長讀變異檢測演算法相比,NanoCaller更具有競爭力,並且可以在複雜的基因組區域中檢測SNP/InDel,有望促進發現更多新的遺傳變異。
參考資料:
Ahsan, M.U., Liu, Q., Fang, L. et al. NanoCaller for accurate detection of SNPs and indels in difficult-to-map regions from long-read sequencing by haplotype-aware deep neural networks. Genome Biol 22, 261 (2021). https://doi.org/10.1186/s13059-021-02472-2