測序基因組涉及從我們從父母那裡繼承而來的約 60 億對核鹼基——即腺嘌呤 (A)、胸腺嘧啶 (T)、鳥嘌呤 (G) 和胞嘧啶 中的一小段 DNA 取樣。基因組測序由兩項關鍵技術實現:“讀取”相對較小 DNA 片段的 DNA 測序儀(硬體),以及結合讀取以識別個體基因組與參考基因組的不同之處和方式的變異呼叫程式(軟體),例如一種是在人類基因組計劃中組裝的。這些變異可能是遺傳疾病的指標,例如患乳腺癌、肺動脈高壓或神經發育障礙的風險升高。
2017 年,我們釋出了 DeepVariant,這是一個開源工具,它使用卷積神經網路(CNN)識別測序資料中的基因組變異。測序過程開始於物理樣本由少數儀器中的任何一個進行測序,具體取決於測序的最終目標。原始資料由基因組重疊片段的大量讀陣列成,然後被對映到參考基因組。DeepVariant 分析這些對映以識別變異位置並將它們與測序錯誤區分開來。
在 2018 年首次釋出後不久,DeepVariant 經歷了許多更新和改進,包括顯著改變以提高全外顯子組測序和聚合酶鏈反應(PCR) 測序的準確性。
我們現在釋出DeepVariant v1.0,其中包含針對所有測序型別的大量改進。DeepVariant v1.0 是我們向PrecisionFDA v2 Truth Challenge提交的改進版本,它在 4 個儀器類別中的 3 箇中獲得了最佳總體準確度。與之前最先進的模型相比,DeepVariant v1.0 顯著減少了廣泛使用的測序資料型別的錯誤,包括Illumina和Pacific Biosciences。此外,透過與UCSC Genomics Institute的合作,我們還發布了一個將 DeepVariant 與 UCSC 的PEPPER方法相結合的模型,稱為PEPPER-DeepVariant,首次將覆蓋範圍擴充套件到牛津奈米孔資料。
測序技術和 DeepVariant
在過去十年中,大部分序列資料是使用Illumina儀器生成的,這些儀器產生短(75-250 個鹼基)和準確的序列。近年來,出現了可以對更長片段進行測序的新技術,包括Pacific Biosciences,它可以產生長達約 15,000 個鹼基的長而準確的序列,以及 Oxford Nanopore,它可以產生長達100 萬個鹼基的讀數,但錯誤率較高。研究人員可能使用的特定型別的測序資料取決於最終用例。
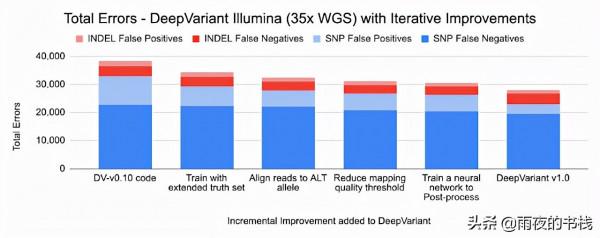
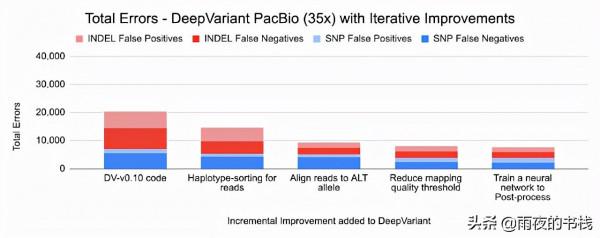
由於 DeepVariant 是一種深度學習方法,我們可以針對這些新的儀器型別快速重新訓練它,確保高度準確的序列識別。準確性很重要,因為遺漏的變異呼叫可能意味著遺漏某種疾病的因果變異,而假陽性變異呼叫可能導致識別不正確的變異。早期最先進的方法可以在 35 倍覆蓋 Illumina 全基因組上達到 ~99.1% 的準確度(~73,000 個錯誤),而早期版本的 DeepVariant (v0.10) 具有 ~99.4% 的準確度(46,000 個錯誤) ,相當於減少了 38% 的錯誤。相對於上一個 DeepVariant 版本 (v0.10),DeepVariant v1.0 將 Illumina 錯誤再減少約 22%,將 PacBio 錯誤再減少約 52%。
DeepVariant 概述
DeepVariant 是一種卷積神經網路 (CNN),它將識別遺傳變異的任務視為影象分類問題。DeepVariant 構造張量,本質上是多通道影象,其中每個通道代表序列的一個方面,例如序列中的鹼基(稱為讀取鹼基)、不同讀取之間的對齊質量(對映質量)、給定讀取是否支援替代等位基因(讀取支援變異)等。然後分析這些資料並輸出三個基因型可能性,對應於存在給定替代等位基因的多少複製(0、1 或 2)。
DeepVariant v1.0 中的技術改進
因為 DeepVariant 對每種資料型別使用相同的程式碼庫,改進適用於 Illumina、PacBio 和 Oxford Nanopore。下面,我們展示了 Illumina 和 PacBio 兩種型別的小變異的數字:SNP(單核苷酸多型性,改變單個鹼基而不改變序列長度)和INDEL(插入和缺失)。
- 在擴充套件的真值集上訓練
該基因在瓶由財團美國國家標準與技術研究院(NIST)建立黃金標準樣品,已知變種覆蓋基因組區域。這些被用作訓練 DeepVariant 的標籤。瓶中基因組使用長讀長技術擴充套件了可信變異集,將標準集描述的區域從基因組的 85% 增加到 92%。這些更困難的區域已經用於訓練 PacBio 模型,並且將它們包含在 Illumina 模型中將錯誤減少了 11%。透過放寬對較低對映質量讀數的過濾器,我們進一步將 Illumina 的錯誤減少了 4%,將 PacBio 的錯誤減少了 13%。
- 長讀長的單倍型排序
我們從母親那裡繼承了一份 DNA,從父親那裡繼承了一份 DNA。PacBio 和 Oxford Nanopore 序列足夠長,可以按親本來源分離序列,這被稱為單倍型。透過向神經網路提供這些資訊,DeepVariant 改進了它對隨機序列錯誤的識別,並且可以更好地確定一個變體是否有來自一個或兩個父母的副本。
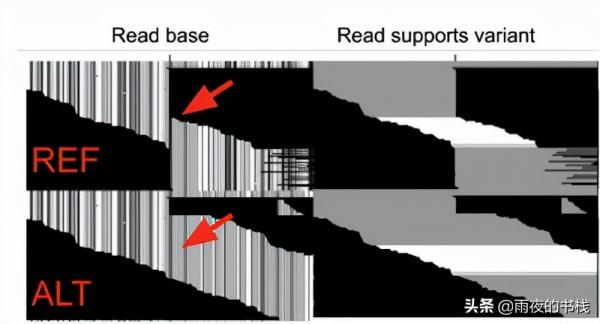
- 將讀取重新對齊到備用 (ALT) 等位基因 DeepVariant 使用已與參考基因組比對的輸入序列片段。如果比對者知道它們存在,那麼包含插入或缺失的變體的最佳比對可能會有所不同。為了捕獲此資訊,我們實施了一個相對於候選變體的額外對齊步驟。下圖顯示了額外的第二行,其中讀取與候選變體對齊,這是一個大插入。您可以看到在第一行突然停止的序列現在可以完全對齊,從而提供更多資訊。
- 使用小型網路對輸出進行後處理
變體可以有多個等位基因,從每個親本繼承不同的鹼基。DeepVariant 的分類器一次只生成一個潛在變體的機率。在以前的版本中,簡單的手寫規則將機率轉換為複合呼叫,但這些規則在某些邊緣情況下失敗。此外,它還將進行最終呼叫的方式與用於訓練網路的反向傳播分開。透過在後處理步驟中新增一個小的、完全連線的神經網路,我們能夠更好地處理這些棘手的多等位基因情況。
- 新增資料以訓練釋出模型
比賽的時間框架被壓縮了,因此我們僅使用與挑戰資料(PCR-Free NovaSeq)相似的資料進行訓練,以加快模型訓練。在我們的產品釋出中,我們尋求多種儀器以及 PCR+ 製劑的高精度。使用來自這些不同類別的資料進行訓練有助於模型泛化,因此我們的 DeepVariant v1.0 版本模型優於提交的模型。
下面的圖表顯示了每項改進所實現的錯誤減少。
訓練混合模型
DeepVariant v1.0 還包括一個用於 PacBio 和 Illumina 讀數的混合模型。在這種情況下,模型利用了兩種輸入型別的優勢,而無需新邏輯。
我們觀察到 SNP 錯誤沒有變化,這表明 PacBio 讀數在 SNP 呼叫方面絕對優越。我們觀察到相對於 PacBio 模型的 Indel 錯誤進一步減少了 49%,這表明 Illumina 和 PacBio HiFi 的 Indel 錯誤模式可以以互補的方式使用。
PEPPER-Deepvariant:使用 DeepVariant 的牛津奈米孔資料管道
在 PrecisionFDA 競賽之前,DeepVariant 模型無法用於牛津奈米孔資料,因為較高的基礎錯誤率為 DeepVariant 創造了太多的候選物件無法分類。我們與加州大學聖克魯斯基因組學研究所合作,該研究所在奈米孔資料方面擁有豐富的專業知識。他們之前訓練了一種名為PEPPER的深度學習方法,可以將候選人範圍縮小到一個更容易處理的數字。DeepVariant 的更大的神經網路然後可以在合理的執行時間下準確地表徵剩餘的候選者。
與牛津奈米孔模型相結合的 PEPPER-DeepVariant 管道是開源的,可在 GitHub 上獲得。該管道能夠在 PrecisionFDA 挑戰中實現優於 DeepVariant Illumina 的 SNP 呼叫準確度,這是第一次有人證明 Nanopore 以這種方式優於 Illumina。
結論
DeepVariant v1.0 並不是開發的終點。我們期待與基因組學界合作,進一步最大化基因組資料對患者和研究人員的價值。