第一篇文章是2022年1月31日發表在Nature biote上的文章“A knowledge graph to interpret clinical proteomics data“,作者用蛋白質組學知識圖譜,提供了從組學資料到輔助臨床決策的可靠的、並經過驗證的分析框架。作者開源了相關資料和程式碼,可以直接遷移到自己的專案中。
將精準醫療應用到臨床決策過程中,取決於整合的多組學資料的情況。但是由於生物醫學資料的質量與多樣性,以及跨不同生物醫學資料庫和出版物中的擴充套件性,對資料整合提出了很高的要求。
作者構建了臨床知識圖譜(CKG),這個開源平臺目前包含了2000萬個節點和2.2億個關係。
圖演算法提供了一個靈活的資料模型,當新的資料庫可用時,該模型很容易擴充套件到新的節點和關係。CKG結合了統計和機器學習演算法,可加速蛋白質組學工作流程的分析和解釋。
透過一組proof-of-concept生物標誌物研究,作者展示了CKG增強和豐富了蛋白質組學資料,併為臨床決策提供了關鍵資訊。
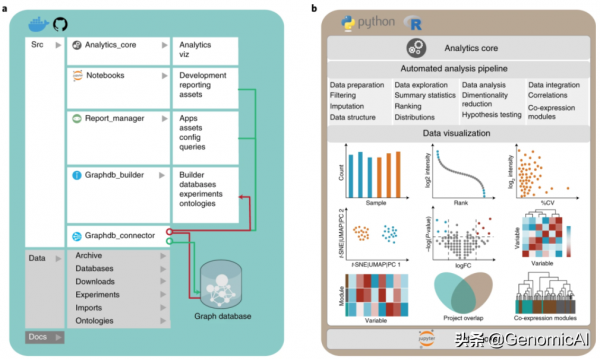
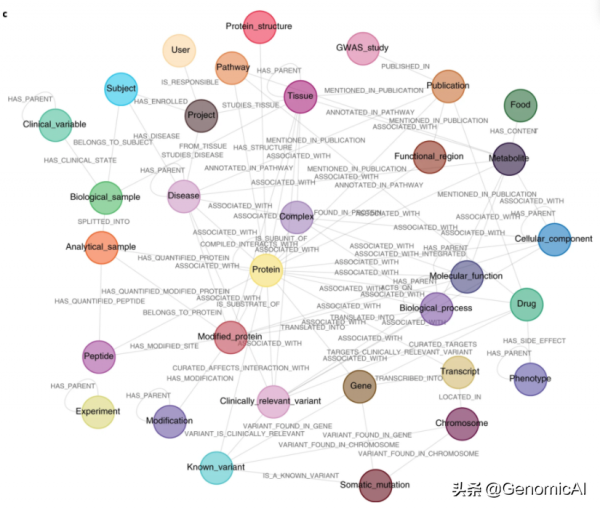
a、CKG架構用python實現,包含幾個獨立的模組:負責連線資料庫(graphdb_builder)、構建圖(graphdb_builder)、分析和視覺化實驗資料(analytic_core)、顯示和啟動多個應用程式(report manager);b、CKG分析核心實現了多種最新的資料科學演算法,用於蛋白質組學的統計分析和視覺化:資料準備、探索、分析和視覺化。c、CKG圖資料庫資料模型旨在整合多層次的臨床蛋白質組學實驗,並用生物醫學資料對其進行註釋。它定義了不同節點(例如,蛋白質、代謝物和疾病)以及連線它們的關係型別(例如,HAS_PARENT和HAS_QUANTIFIED_PROTEIN).
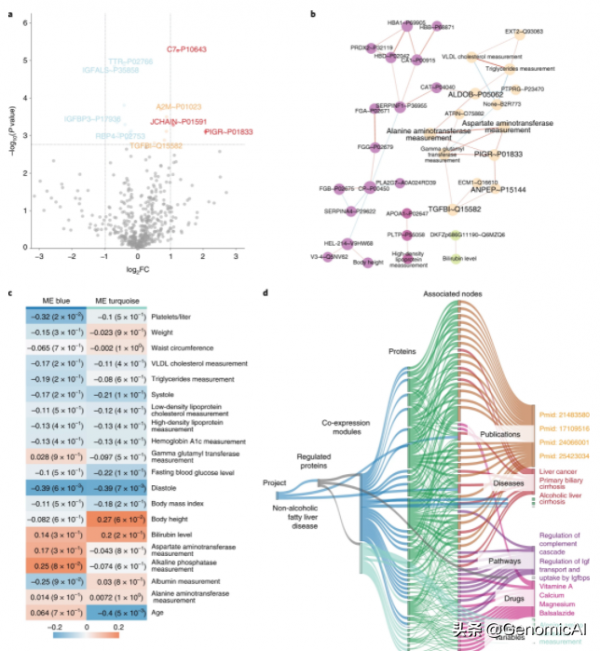
自動CKG分析用於肝病生物標誌物發現
CKG的自動化分析流程復現了(Niu等人的結果[41])之前的結果。
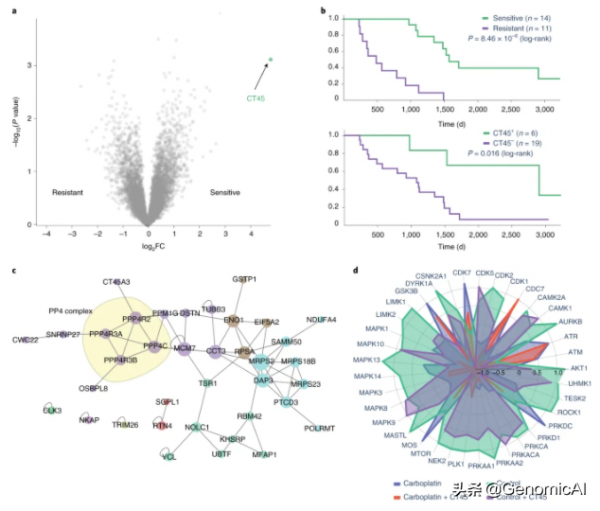
CKG支援多蛋白質組學資料整合,用於癌症生物標誌物的發現和驗證
a , CKG 強調 CT45 是唯一在比較化療耐藥和化療敏感患者的卵巢腫瘤組織時顯著調節的蛋白質 ( n = 25; SAMR s0 = 2; BH FDR < 0.05) (資料來自 Coscia 等人3 ) . b,CKG 的分析管道估計了臨床組敏感和耐藥(雙邊對數秩檢驗)的生存函式,相應的高(前 25%)和低(剩餘 75%)CT45 表達,並確認明顯更長的疾病-高 CT45 表達組的自由生存。C, 相互作用蛋白質組學揭示了 PP4 磷酸酶複合物的亞基作為 CT45 的直接相互作用物,CKG 將其顯示為 PPI 網路中的簇,確認已知的相互作用物並突出潛在的新相互作用物(由簇著色的節點)。d,CKG 中的磷酸蛋白質組學分析確定了顯著調控的位點,並將它們與上游激酶調節劑聯絡起來。在這些激酶調節劑中,CDK7、CDC7、ATR 和 ATM 受卡鉑作用的影響很大。FC,摺疊變化。
程式碼:https://github.com/MannLabs/CKG
第二篇論文是2022年2月11日發表在Nautre comm上的文章“Machine learning prediction and tau-based screening identifies potential Alzheimer’s disease genes relevant to immunity”。
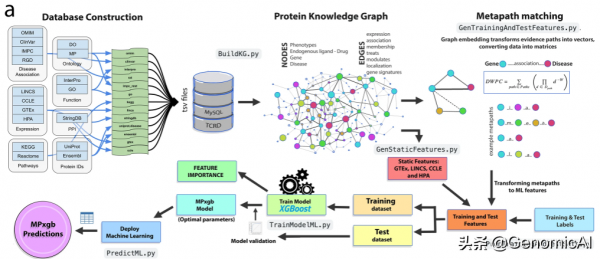
為基於元路徑的機器學習開發了(ProteinGraphML)知識圖譜。
作者基於Target Central Resource Database 蛋白知識圖譜和透過元路徑匹配將證據路徑轉換為向量,然後提取了基因和疾病之間的特徵,最後使用稱為MPxgb(AD)的XGBoost訓練和優化了模型。
基於該知識圖譜分析確定了潛在的AD(老年痴呆)風險基因:FRRS1, CTRAM, SCGB3A1, FAM92B/CIBAR2, and TMEFF2. FRRS1 and FAM92B被認為是dark genes,但是TREM2-TYROBP, IL-1β-TNFα, and MTOR-APP是風險基因,提示與AD的發病機制相關。
作者透過實驗驗證了預測的生物標誌物的準確性:
(1)在人類死後 AD 大腦中,前 20 個 MPxgb(AD) 預測基因中有 5 個在 mRNA 水平上發生了改變,9 個在蛋白質水平上發生了改變。
(2)siRNA 介導的CRTAM、FOXP4、GRIN2C、LILRA3、PIBF1、SCGB3A1和TXNDC12 敲除減少炎症誘導的 tau 磷酸化。
(3)一些底部 MPxgb(AD) 預測基因顯示 AD 相關性,但與免疫/炎症無關。
相關分析表明,預測的前 20 個 MPxgb(AD) 基因中有兩個(SCGB3A1 和 CRTAM)與免疫相關。
第三篇文章是2022年1月23日發表在ICLR2022上的文章“OntoProtein:Pro
-tein Pretraining with Gene ontology embedding”。
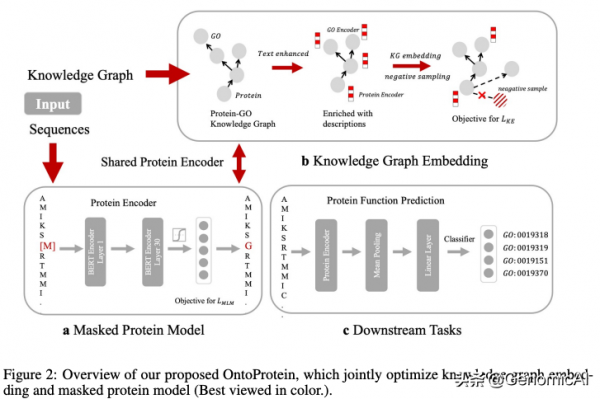
作者將GO(基因本體)中的結構用於蛋白質預訓練模型的通用框架,構建了一個新的大規模知識圖譜,該知識圖譜中的所有節點由GO及其相關蛋白質組成,基因註釋文字或蛋白質序列描述。
OntoProtein基於基因本體嵌入的蛋白質預訓練模型,是第一個將外部知識整合到蛋白質預訓練中的通用框架。受生物特徵機制的啟發,作者設計了一種知識感知的負取樣策略。
實驗結果表明,OntoProtein作為基因本體嵌入的蛋白質預訓練語言模型超越了最優的模型,並在蛋白質-蛋白質相互作用和蛋白質功能預測方面比基線模型產生了更好的效能。
該蛋白質預訓練模型超過了基線模型TAPE。
參考文獻:
[1] A knowledge graph to interpret clinical proteomics data
[2] Machine learning prediction and tau-based screening identifies potential Alzheimer’s disease genes relevant to immunity
[3] OntoProtein:Protein Pretraining with Gene ontology embedding
[4] Plasma proteome profiling discovers novel proteins associated with non‐alcoholic fatty liver disease