近年來,誘導多能幹細胞衍生的類器官,為研究人體器官發育提供了模型。據悉,單細胞轉錄組學能對這些系統中的細胞狀態進行高度解析。然而,此前尚無方法去直接測量血統關係。
基於此,浙大校友何志嵩和同事們共同研發了譜系記錄儀 iTracer,它能將報告條形碼與可誘導的 CRISPR–Cas9 瘢痕相結合,並與單細胞和空間轉錄組學相容,藉此即可探索大腦類器官發育過程中的克隆性和譜系動力學。
詳細來說,該記錄儀允許從初始化 iPSC 池進行克隆追蹤,同時還可以使用誘導性疤痕在不同時間點進行譜系記錄,既可以進行克隆分析,也可以探索細胞命運建立的時間動態,規避了多輪誘導性疤痕標記的低效問題。
2021 年 12 月30日,相關論文以《人腦類器官中的譜系記錄》(Lineage recording in human cerebral organoids)為題,發表在 Nature Methods 上[1]。
長期以來,何志嵩所在研究組對人類早期胚胎髮育時中樞神經系統尤其是腦部發育、神經發育相關疾病的發病機理、以及發育時的表現有著濃厚興趣。
研究中,他們使用大腦類器官微型三維組織培養技術,定向誘導人類胚胎幹細胞,或誘導多能幹細胞分化成不同型別的腦部神經元以及其他細胞型別,從而對人類腦部早期發育過程、進行模擬和機理研究。
為全面描繪這一過程中細胞分子特徵的變化,他們大量使用單細胞測序技術、尤其是單細胞轉錄組技術,來獲取期間可能出現的動態細胞狀態,並透過整合不同時間點的樣本資料、以及各種計算分析手段,來推測不同細胞型別的發生過程。
不過這一方法也有侷限性,當前單細胞測序技術需要對細胞進行破壞式測量,因此只能獲取細胞在最終測量時、關於細胞狀態的單一快照。
這樣既無法得知細胞的歷史狀態,更無法得到細胞的譜系資訊,也就是說這些細胞系在特定時間由同一個幹細胞分裂分化所產生的後代細胞。
而對於更好地描繪大腦早期發育過程,這些資訊至關重要。為此,何志嵩和他的同事們需要一種新技術,在相容當前單細胞測序技術的同時,可進一步獲取細胞譜系資訊,即進行細胞譜系追蹤。
當前,可相容單細胞轉錄組測序的細胞譜系追蹤技術,主要分成兩類:
一類為靜態序列標記,即構建一個高複雜度的條碼序列文庫、轉染並整合到幹細胞中,從而標記不同的幹細胞;
另一類為基於 CRISPR 技術的動態序列標記,即透過誘導 CRISPR 編輯系統,對一個特定序列引入一系列隨機突變,並透過每個幹細胞中檢測到的突變組合,來構建細胞譜系資訊。
這兩種技術各有利弊:靜態序列標記僅能獲取單個時間點的譜系資訊;而基於 CRISPR 技術的動態標記,則存在比較嚴重的標記衝突問題,多次獨立編輯事件會產生同樣的突變,從而導致重構譜系資訊的不準確。
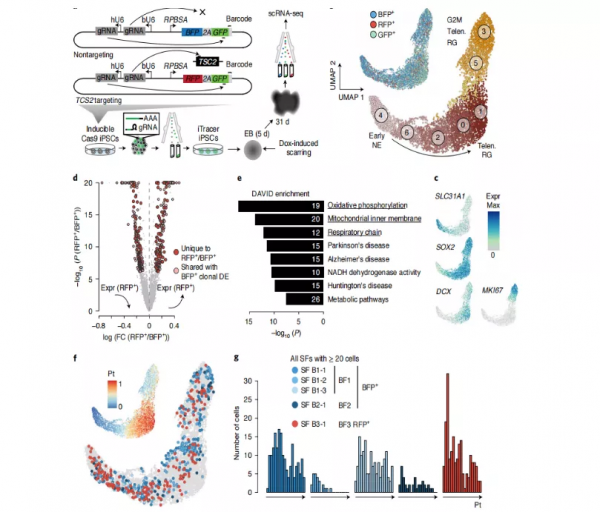
為更好地解決這一問題,何志嵩和他的同事一起整合了這兩類技術,開發出 iTracer 技術。iTracer 包含靜態序列標記原件,可用於標記起始時間點的不同幹細胞,也包括基於 CRISPR 編輯系統的動態序列標記,結合帶有可誘導 Cas9 蛋白基因的幹細胞,即可在特定時間點產生額外的隨機突變,從而得到第二層細胞譜系資訊。
概括來說,該技術綜合了兩類已有技術的優點,既能獲得多個時間點的譜系層次資訊,也能降低標記衝突問題造成的影響。
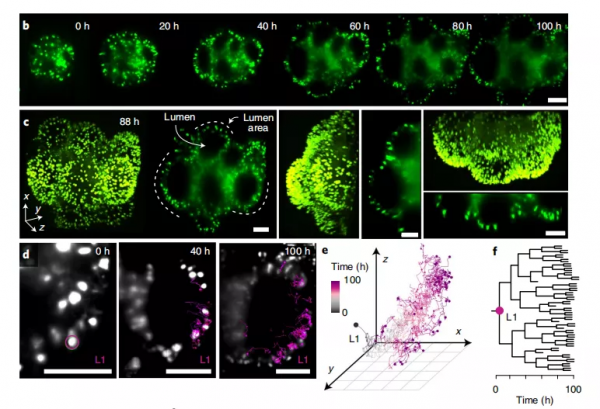
隨後,他們將 iTracer 技術用於大腦類器官系統中,藉此研究大腦類器官中不同型別神經元之間的譜系關聯。
他發現,代表不同腦區的神經元,傾向於由不同的多能幹細胞產生,且同一多能幹細胞產生的後代細胞,在空間分佈上呈現出聚集分佈的特徵。
這一現象暗示著,在分裂分化過程中,大腦類器官的細胞並未發生顯著的細胞遷移,因而其後代細胞呈聚集分佈,並在類似的微環境作用下,被誘導為同樣型別的神經元。
透過使用長時間光片顯微鏡技術,對稀疏核標記的大腦類器官進行追蹤觀察,這一假設也得到了進一步證明。
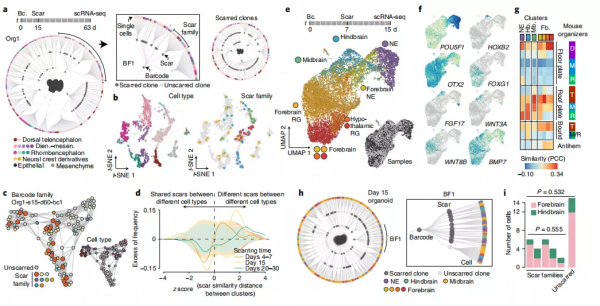
而在此基礎上,透過在不同時間點引入動態序列標記,還可得到大腦類器官中不同細胞型別、特別是不同型別神經元的命運決定關鍵時間點,並對同一多能幹細胞產生的不同後代神經元的分化情況進行比較。
此外,透過在 iTracer 的基礎上額外引入一個針對靶點基因的 gRNA,還能在利用 iTracer 記錄細胞譜系資訊的同時,對靶點基因進行基於 CRISPR 技術的基因敲除,並據此對基因敲除如何影響細胞譜系的發展分化進行研究。
據介紹,何志嵩和同事們也將 iTracer-perturb 這一技術用於大腦類器官中,並就 TSC2 基因對大腦類器官中神經元發育的影響,進行了初步探討。
結合基於CRISPR技術基因敲除的iTracer技術
該研究始於 2017 年。何志嵩所在小組主要關注的生物系統,一般和組織發育、組織再生、幹細胞分化等相關,其中包含大量細胞狀態的動態轉換。
因此,彼時已逐漸成熟的大規模單細胞轉錄組測序技術,讓他得以研究這些動態過程。但是,受限於單細胞測序技術對所測量細胞的破壞性,如何透過實驗手段直接獲得不同細胞之間的譜系發生關係,成為何志嵩和他的同事們迫切希望解決的問題。
當時,包括華盛頓大學遺傳學和發育生物學系教授薩姆·莫里斯(Sam Morris)團隊的靜態序列標記技術 CellTag,以及德國柏林醫學系統生物學研究所(BIMSB)揚·菲利普·容克(Jan Philipp Junker)團隊的動態序列標記技術 LINNAEUS 等剛剛出現。
它們都是可相容單細胞轉錄組測序技術的細胞譜系追蹤技術,何志嵩所在的研究團隊也開始進行使用。但在隨後,他們很快就發現了上述技術的侷限性,因此想開發一種能將靜態序列標記和動態序列標記結合的方法。
這一想法變成課題並於 2018 年正式開始,他們先是構建了包含 iTracer 元件的轉座子質粒載體,並開始在大腦類器官中利用 bulk 測序論證 iTracer 技術的可行性。
在確定技術可行後,在 2019 年起他們將 iTracer 技術用於大腦類器官系統中,藉此獲得了在不同時間點進行動態序列標記誘導的大腦類器官單細胞轉錄組測序資料,並進行資料分析。
2020 年初,迎來資料分析的初步結果。何志嵩藉此得知,多能幹細胞在分裂分化、以及產生大腦類器官的過程中,並未發生明顯遷移。基於此,他做出了同一譜系的細胞在空間中出現聚集的假設。
為證明該假設的合理性,一方面他和研究組的其他成員一起結合 iTracer 技術與空間轉錄組,去觀察不同細胞譜系在大腦類器官中的分佈;另一方面他們還使用長時間光片顯微鏡技術,對大腦類器官的早期發育進行記錄,並對其中數個被進行細胞核標記的幹細胞、及其後代細胞進行追蹤。
這兩個方法均驗證了上述假設。然後,透過比較在不同時間點引入動態序列標記的樣本,何志嵩對大腦類器官中、不同神經元的“命運發生”重要時間節點做以推斷,並透過產生對應時間的大腦類器官的單細胞轉錄組資料,確認了在該時間點中不同神經祖細胞的存在。
考慮到此次使用的 10x Genomics 的單細胞測序技術,實際上只測量了每個大腦類器官中的極個別細胞,這導致每個細胞譜系只能獲得少數幾個細胞的資料,也給譜系之間的直接比較帶來困難。
為此,研究團隊還對單個類器官進行顯微切割,並對所得到的各個部分,分別進行了深度的單細胞轉錄組測序,從而實現了對細胞譜系的充分取樣。
最後,他們又對 iTracer 技術做了拓展,在原來 iTracer 載體的基礎上再引入一個靶向目標敲除基因的 gRNA,從而將它和基於 CRISPR 技術的基因敲除結合起來。
這讓他們對所得到的 iTracer-perturb 技術進行了初步應用,並對 TSC2 基因在大腦類器官發育過程中的作用、以及對細胞譜系的潛在影響進行了研究。
可用於所有體外細胞培養系統
研究中還曾發生過一個小插曲,何志嵩表示:“對於不同神經元富集不同多能幹細胞譜系的這個結果,一開始讓人很是始料未及。因為我們認為,用於培養大腦類器官的多能幹細胞是均一的,所以很自然地覺得,不同幹細胞會以均等的可能性,產生各種神經元。”
因此,當時他的第一反應是分析方法可能有問題,或由於某些原因導致不同幹細胞,確實存在產生不同神經元的傾向性。
直到在一次討論中,他才突然意識到其實這個結果完全可能用幹細胞低遷移性導致的細胞譜系非均勻分佈,以及不同神經元的空間非均勻分佈來解釋。
為進一步確認該想法,他又設計了 iTracer 與空間轉錄組學的結合、以及長時間光片顯微鏡的實驗,而這些實驗又確確實實地證實了上述想法。
對於潛在應用,他表示:目前該技術的應用,應該會停留在基礎研究領域,但絕不侷限於大腦類器官這一個系統。理論來看,iTracer 技術可用於所有體外細胞培養系統,唯一要求是所使用的幹細胞必須帶有可誘導的 CRISPR 編輯系統。”
生於廣東,本科就讀於浙大生命學院
何志嵩是廣東中山人,生於 1987 年。本科就讀於浙江大學生命科學學院生物資訊學專業,2009 年保送至當時的中科院上海生命科學研究院的馬普學會-中科院計算生物學夥伴研究所,師從菲利普·海托維奇(Philipp Khaitovich)研究員。
2015 年博士畢業後,他繼續留組研究至 2017 年。在中科院期間,何志嵩以第一作者或通訊作者身份在 Nature Neuroscience、Molecular Psychiatry 等期刊發表了數篇論文。
2018 年,他來到德國萊比錫馬普學會進化人類學研究所,並加入芭芭拉·特魯特林(Barbara Treutlein)教授的單細胞基因組學研究組,進行博後研究。
2019 年,他隨團隊搬到瑞士蘇黎世聯邦理工學院生物系統科學與工程系,並繼續在特魯特林團隊進行博後工作。
期間,何志嵩的研究方向是單細胞功能基因組學、特別是單細胞轉錄組學資料的分析及方法開發,並以大腦類器官及其他類器官系統作為模型,研究早期人類大腦和其他器官發育過程中細胞命運的決定機理。
來到瑞士後,他先後以一作或通訊作者身份在 Nature、Genome Biology 及Stem Cell Report 中發表論文。
2021 年 7 月份,何志嵩成為團隊的高階研究助理,在做科研的基礎上,開始負責計算資源管理、以及部分學生指導工作。
對於此次論文的後續研究,他表示:“我們正將 iTracer 技術用於組內的幾個其他研究,並已取得不錯的結果。同時,也希望對 iTracer 做進一步改進,主要會研究 CRISPR 系統引入動態序列標記的效率較低的問題。”
-End-
參考:
1、He, Z., Maynard, A., Jain, A. et al. Lineage recording in human cerebral organoids. Nat Methods 19, 90–99 (2022). https://doi.org/10.1038/s41592-021-01344-8