華中科技大學寧康教授團隊基於地球上4個主要生物群落(腸道、湖泊、土壤和發酵罐)的42.5億個微生物群落序列構建了AI模型MetaSource,預測出了1,044個未知結構蛋白的可靠結構。
背景介紹
目前,蛋白三維結構預測主要有兩種策略:一種是基於模板的建模,以已解決的結構為模板構建結構模型,需要在PDB中有同源模板;第二種是無模板建模(FM)方法(或從頭開始建模),專注於對PDB中沒有密切同源結構的硬蛋白進行建模。最有效的FM方法,包括Rosetta、QUARK和I-TASSER,以及最近的AlphaFold和trRosetta,依賴於先驗空間約束,透過深度神經網路學習,從基於同源序列的多序列比對(MSAs)的共進化資訊中獲得。因此,要對“硬”蛋白進行三維結構建模,需要有足夠數量的同源序列,以保證深度機器學習模型的準確性和後續三維結構構建的質量。
近年來,人們在利用宏基因組序列資料來增強MSA和FM模型的構建上付出了大量努力。最近,Wang等人研究了Tara Oceans微生物基因組的有效性,發現微生物基因組可以為高質量的MSA構建以及蛋白結構和功能建模提供額外的幫助。這一結果表明,微生物組序列在FM結構摺疊和基於結構的功能註釋中發揮了重要作用,這些序列代表著地球上最大的微生物物種之一。
儘管宏基因組輔助的三維結構建模取得了成功,但仍有成千上萬的Pfam家族的建模結構不能令人滿意。一個關鍵的原因是,儘管序列積累迅速,但目前的序列資料庫還很不完整,許多FM目標的同源序列非常少。另一方面,微生物組資料庫已變得非常龐大,這使得全面且平衡的資料庫搜尋變得越來越緩慢和困難。因此,一種具有精確MSA構造的均衡序列挖掘方法對於提高序列資料庫搜尋效率和後續三維結構建模具有至關重要的意義。
主要內容
華中科技大學生命科學與技術學院寧康教授團隊聯合美國密歇根大學計算醫學與生物資訊系張陽教授團隊,假設在微生物生態位(生物群落)和蛋白質家族之間存在著一種內在的進化聯絡,依此聯絡建立一種有針對性的方法,可以提高MSA構建和蛋白質結構預測的效率和準確性。為了檢驗這一假設,研究者從EBI宏基因組資料庫(MGnify)中收集了一個包含42.5億個微生物組序列的模型庫,該資料庫覆蓋了4個主要的生物群落。“邊際效應”分析表明,特定生物群落對不同Pfam家族同源序列的補充作用存在顯著差異。隨後,研究者開發了一個名為MetaSource的機器學習模型來預測靶蛋白的源生物群落,該模型可顯著提高接觸圖和三維結構模型的精度,同時節省了超過三倍的計算機記憶體和CPU時間。相關的研究成果以 “Decoding the link of microbiome niches with homologous sequences enables accurately targeted protein structure prediction” 為題釋出在國際著名期刊Proceedings of the National Academy of Sciences of the United States of America上。
C-I-TASSER模型、Pfam家族結構未知的MSAs以及基準資料集下載地址
https://github.com/HUST-NingKang-Lab MetaSource /releases
微生物群落樣本包含來自數千個屬的數十億個不同功能基因
研究者從4個典型的微生物生態位(圖1A)中,採集了1705個微生物組樣本。從這些生物群落中共預測了42.5億個蛋白質序列(功能基因),鑑定出169個門8721個屬,圖1B-D展示了生物群落特異性的分類剖面。這些結果表明,微生物群落中存在著不同的微生物群系,進一步的研究揭示了微生物群落分類剖面與其活菌群之間的相關性:在腸道菌群中,如厚壁菌門(平均相對丰度:0.41±0.28)和擬桿菌門(平均相對丰度:0.26±0.14)為優勢門。
圖 1. 不同微生物組樣品的分類和功能分析。圖片來源於PNAS
在從這4個生物群落中獲得的42.5億個蛋白質序列中,研究者觀察到了生物群落特異性的功能圖譜。與分類圖譜相似,4個生物群落中存在不同的功能註釋(圖1E):僅在腸道生物群落中就檢測到3.6億個(68.4%)註釋,湖泊、土壤和發酵生物群落中分別檢測到0.038億個(29.9%)、0.32億個(62.7%)和0.016億個(24.2%)註釋。基於功能剖面的PCoA結果顯示:這四個生物群落之間存在明顯差異(圖1F)。
宏基因組來源的蛋白助力上千蛋白家族無需同源模板即可成功結構建模
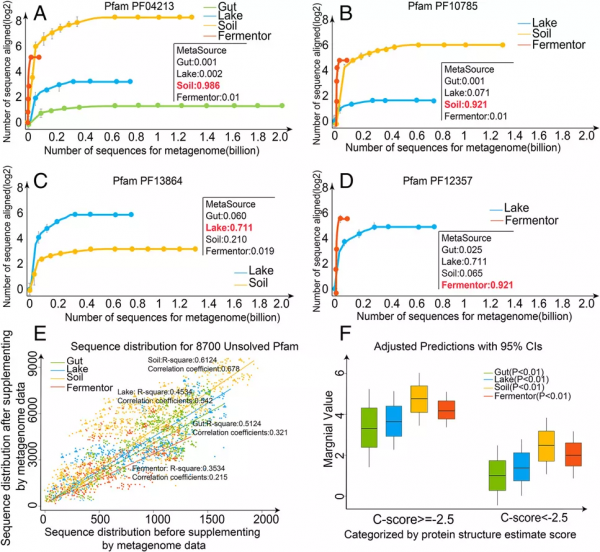
研究者從8700個結構未知的Pfam家族中,選取了2214個MSA >16 (= 24) 的Neff家族。從微生物組序列開始,將深度學習C-I-TASSER方法擴充套件到2214個結構未解決的Pfam家族來構建相應模型。基準測試結果顯示,置信度(C-score)為-2.5的模型通常具有正確的摺疊,C-I-TASSER發現Pfam家族中有47%(1044 / 2214)是可摺疊的(圖2A)。圖2B是C-I-TASSER模型在2214個未知Pfam家族上的C-score直方圖分佈。圖2C顯示了C-score > -2.5的13個Pfam家族。雖然大多數靶標摺疊正確,但有兩個案例(PF3864和PF12357) TM-評分在0.5以下。
圖2. 未知Pfam Hard家族的結構建模結果。圖片來自PNAS
不同生物群落中同源序列的富集
C-I-TASSER預測可摺疊的1044個Pfam家族中可以觀察到從特定生物群系中富集的同源序列,在一個生物群系中可識別出964個Pfam家族(964/ 1044,92.3%),其Neff值大於其他三個生物群系,其中Gut為105戶,Lake為116戶,Soil為617戶,Fermentor為126戶(圖3A)。
為了揭示已解決的Pfam家族在其生存環境中的重要作用,研究者對964個具有單個對應生物群系的Pfam家族分別進行了分類剖面和功能組成分析。結果表明了微生物群落序列對Pfam結構建模的特異性貢獻(圖3B)。與微生物群落樣本相似, 964個Pfam家族的物種異質性分佈反映了生物群落特異性的富集模式。此外,特定Pfam家族的優勢種往往是MSA構建輔助微生物組樣品的優勢種。例如,在Pfam家族中標記了腸道生物群系(圖1B和3B), Firmicutes和Bacteroidetes(均屬於腸道)是Pfam家族的優勢門(分別為0.41±0.28和0.26±0.14)和相應的源生物群系(分別為0.48±0.31和0.31±0.15),說明這種生物特異性富集模式受微生物群落組成的影響。
除結構建模外,964個Pfam家族的功能組成也為這種生物群落特異性的富集模式提供了有用的見解。例如,基於GO註釋,368個Pfam家族被對齊到GO level-3 Biological Process(286)、Molecular Function(90)和Cellular Component(189)(圖3D-F)。
圖 3. 基於C-I-TASSER的可摺疊Pfam家族的分類和功能特性。圖片來源於PNAS
邊際效應分析揭示生物群落-序列-Pfam關係
研究者列出了四個生物群落對所有8700個未知的Pfam家族的邊際效應;資料表明,不同生物群落對某一特定Pfam的貢獻可能存在很大差異,這可以從它們的ME值中得到反映。在圖4 A-D中,研究者展示了生物群落對PF04213、PF10785、PF13864和PF12357四個樣本的MSA收集的貢獻,其中微生物群落樣本被隨機分配到不同序列號的MSA收集中。對於不同的Pfam家族,序列同源庫主要由不同的生物群落資料集組成,這再次表明在同源序列補充方面,生物群落和Pfam之間存在很強的聯絡。
在圖4F中,研究者根據C-I-TASSER摺疊結果,將Pfam家族分為兩組。結果表明,C-score ≥ -2.5分家族的ME值明顯高於C-score < -2.5分家族(t檢驗結果為5.27±3.44 vs 1.28±0.85, P值= 3.86e-26)。因此,邊際效應值也與生物群系特異性宏基因組序列透過補充更多的同源序列來輔助三維結構組裝模擬的能力密切相關。
圖 4. Pfam家族的邊際效應評價。圖片來源於PNAS
有效同源序列補充的MetaSource預測模型
研究者開發了MetaSource預測模型來識別一個或一組生物群系,可以更好地補充特定Pfam家族的同源序列集合。
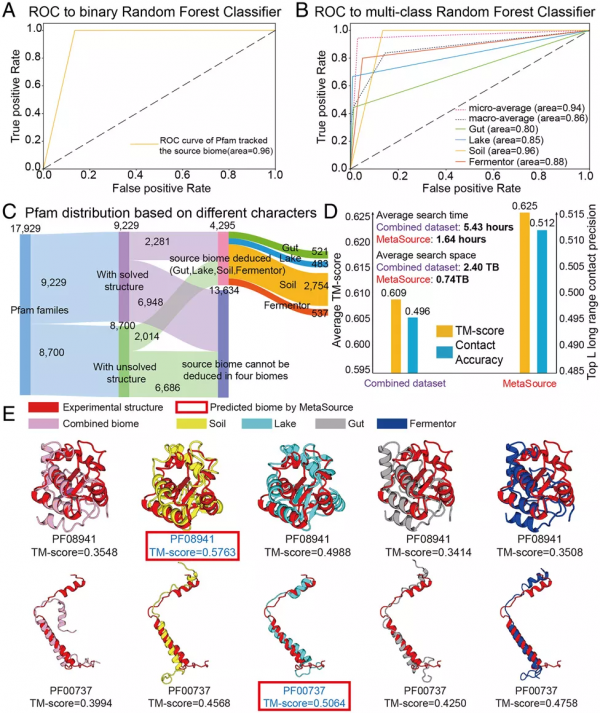
首先,為了確定查詢的Pfam家族的源生物群系是否為四個生物群系之一,研究者以單個生物群系標記的964個Pfam家族為訓練集,7736個結構未知Pfam家族為測試集構建了二值分類模型。如圖5A所示,在二元分類檢驗中,MetaSource在0.001個排列P值下的AUC為0.96。
其次,採用多分類隨機森林演算法來預測Pfam家族的四個生物群系中最可能的源生物群系。964個Pfam家族經過20次交叉驗證迭代,結果顯示MetaSource對Pfam家族具有很強的預測能力,在0.001排列P值下平均AUC為0.94。
圖 5. MetaSource對Pfam家族預測的源生物群系。圖片來源於PNAS
為了進一步研究Metagenome資料庫和MetaSource模型在3D結構建模中的實際用途,研究者將204個已知結構的Pfam家族納入驗證集。首先,C-I-TASSER利用基因組資料庫中的MSA (DeepMSA的step 2結果)生成了TM-score = 0.583的模型,該模型僅使用Uniclust30基因組資料庫,比C-I-TASSER模型高2.5%。
在DeepMSA的第3步新增宏基因組資料庫後,C-I-TASSER模型的TM-score提高到0.609,比僅使用P值為3.8E-11的基因組資料庫高出4.5%。這一結果,透過擴充套件MSA的覆蓋範圍和多樣性,再次證明了宏基因組資料庫在三維結構預測中的有效性。總的來說,透過將DeepMSA工具與宏基因組資料庫相結合,C-I-TASSER模型的TM-score比簡單的一步HHblitMSA收集方法提高了7%。
MetaSource能夠預測獲得最高Neff(或最高TM-score)的生物群落,在204個已知的Pfam家族的準確率為79.9%(或80.2%)(排列P值:0.001)。在圖5D中,研究者進一步比較了C-I-TASSER模型。結果表明,雖然序列資料庫的容量要小得多,使用Meta-Source的目標資料集的接觸精度(0.512)和TM-score(0.625)卻高於組合資料集(0.496和0.609)。
在圖5E中,研究者給出了PF08941和PF00737兩個已知結構的Pfam家族,MetaSource預測土壤和湖泊分別為最佳源生物群落。在這兩種情況下,只有使用MetaSource預測生物群系的模型,才能建立一個TM-score高於0.5的模型。
總結
本研究利用2.4TB微生物組測序資料模型庫,在更加宏觀的層面對微生物組-序列-蛋白結構之間的關聯性做了驗證,並構建了機器學習模型來更高效地利用微生物組輔助預測蛋白結構及功能。
本研究僅以C-I-TASSER結構建模方法作為概念證明,更多的基因組資料集,包括其他生態指標,可在未來直接納入這個模型。此外,隨著該領域的快速發展,僅考慮接觸圖約束的C-I-TASSER,可能不再代表蛋白結構預測的技術水平。
該研究首次發現了微生物組的同源序列在不同生態位中的不均衡分佈和富集特徵,相關領域人員可以藉此更深入地理解功能基因的適應性進化。靶向宏基因組選擇方法能夠對蛋白結構預測和功能註釋的效率和有效性產生更加顯著的影響。
參考文獻
Cihan Ayaz, Lucas Tepper, Florian N. Brünig, Julian Kappler, Jan O. Daldrop, Roland R. Netz, Non-Markovian modeling of protein folding, Proceedings of the National Academy of Sciences Aug 2021, 118 (31) e2023856118; DOI: 10.1073/pnas.2023856118.
中大唯信頭條號與中大唯信公眾號、唯信計算訂閱號均由中大唯信科技有限公司運營,歡迎關注轉發,未經授權禁止轉載