隨著小麥基因組的不斷完善與提高,小麥分子育種與功能基因組學研究進入新的時期。相對於 Affymetrix 與 Illumina SNP晶片,液相捕獲晶片不僅能夠提供靈活設計合成方案,提供更為豐富的基因組變異資訊。同時由於規模化生產及測序成本的下降,利用液相捕獲晶片的群體測序方案價格已經遠低於固態SNP晶片。
天成未來(Tcuni)聯合國內多家單位開發了多款小麥捕獲測序晶片。基於前期構建的大規模基因型資料庫在2000份材料中篩選出高質量的多型性位點設計合成了45K、60K、120K、160K等不同密度的晶片。基於“矮抗58”及“科農9204”等基因組設計合成了現代品種更為適用的全外顯子捕獲晶片(根據Wheat 10+ Genomes Project研究發現每個現代品種相對於中國春有3%~4%基因數量差異(Walkowiak et al., 2020),使用中國春參考基因組的探針無法全面覆蓋這些資訊。)。
公司的捕獲晶片參與了國內多個大規模自然群體/突變體群體的測序。其中包括與中國農科院賈繼增團隊、河南科技學院茹正剛團隊等國內單位完成的國內首個有參考基因組(“矮抗58”)突變體庫測序,以及煙臺大學宋建成團隊完成的國內首個商業化突變體庫(“濟麥22”)。同時公司的捕獲測序被應用於數千份材料的關聯分析(GWAS)、基因定位(BSA)、標記開發、種質資源鑑定等工作。
近期公司推出了第一款小麥泛基因組SNP晶片TA-PanGen-150K。在中國春2.1版本HC基因的基礎上,增加Wheat 10+ Genomes Project、Fielder、AK58、JM22、YZ4110等國內外基因組有代表性的基因片段。大量現代品種特異基因片段的加入,使得該SNP晶片能夠更好的應用於現代品種材料的基因分型、遺傳定位等工作。

圖2 小麥泛基因組SNP晶片設計示意圖。(圖中藍色線條代表基因,橙色豎線代表SNP位點,綠色線條代表捕獲測序的區域。)
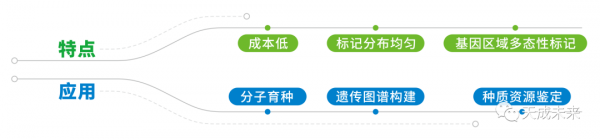
TA-45K|45K晶片
使用近2000份小麥重測序及外顯子測序資料分析,覆蓋國內大部分地區的小麥育成品種。選取基因組上勻分佈相對均的4.43萬個高質量的多型性區域,包含超過12萬個多型性位點,所有位點均位於基因區域。基因組平均每500Kb一個多型性區域,一個多型性區域包含2~3個SNP位點。
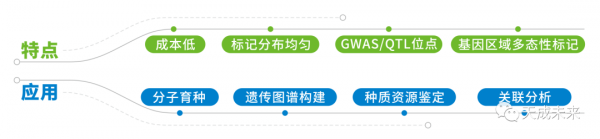
使用近2000份小麥重測序及外顯子測序資料分析,覆蓋國內大部分地區的小麥育成品種。選取基因組上勻分佈相對均的6.1萬個高質量的多型性區域,包含超過15萬個多型性位點。基因組平均每300Kb一個多型性區域。在45K基礎上增加了大量已報道的GWAS/QTL位點。
使用近2000份小麥重測序及外顯子測序資料分析,覆蓋國內大部分地區的小麥育成品種。選取位於基因組上相對保守區域的12萬個多型性區域,包含20萬個多型性位點。所有多型性區域位於基因及上下游2K範圍內。
使用近2000份小麥重測序及外顯子測序資料分析,覆蓋國內大部分地區的小麥育成品種。選取位於基因組上非保守區域的16萬個多型性區域,包含40萬個多型性位點。所有多型性區域位於基因及上下游2K範圍內。
在小麥基因組2.1版本釋出後(Zhu et al., 2021),我們在中國春1.1版本基礎上(Appels et al., 2018),進行產品升級。該晶片包含全部的HC基因及5萬個在2000份轉錄組資料中高表達的LC基因。
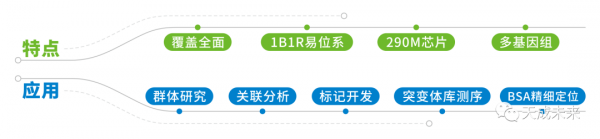
與中國農科院賈繼增團隊、河南科技學院茹正剛團隊等國內單位合作,基於國內品種“矮抗58”參考基因組進行晶片的設計開發與合成。WES-AK58晶片是目前國內覆蓋基因區域最大的外顯子晶片,包含基因CDS區域及啟動子區域。由於“矮抗58”為小麥1B/1R易位系材料,所以基於此基因組開發的外顯子探針WES-AK58能夠很好對國內具有1RS片段的材料進行捕獲測序。同時因為加入了中國春的1BS、偃展4110、濟麥22等材料的基因序列,對非1B/1R易位系材料也能有很好的資料表現。
與中科院遺傳發育研究所凌宏清團隊合作,基於國內品種“科農9204”參考基因組設計合成的全外顯子晶片。晶片包含全部HC基因的CDS區域及部分LC基因CDS序列。由於探針覆蓋集中在CDS區域,能夠在較少的資料量情況下有著較高的覆蓋深度,對於大量樣品的測序成本較低。同時因為技術的提升,該款晶片在低測序數量上的均一性上有著非常好的表現。

TA-WES-Fielder | Fielder全外顯子晶片
Fielder作為使用最為廣泛地遺傳轉化材料(Sato et al., 2021),在功能基因組學有著非常重要的地位。同時Fielder參考基因組也是目前公開使用的組裝最完整小麥基因組。為了更好的服務於小麥功能基因組研究,我們開發了基於Feilder材料的外顯子晶片。
TA-Custom Panel | 個性化基因晶片定製
公司提供個性化的晶片定製合成,開發專屬的SNP晶片、基因家族晶片、育種檢測晶片等。具體技術細節請與我們聯絡。
產品與技術支援:
電話:028-85923313
微信公眾號:
Reference:
Appels, R., Eversole,K., Feuillet, C., Keller, B., Rogers, J., et al. 2018. Shifting the limits inwheat research and breeding using a fully annotated reference genome. Science,361(6403). https://doi.org/10.1126/science.aar7191.
Hao, C., Jiao, C.,Hou, J., Li, T., Liu, Hongxia, et al. 2020. Resequencing of 145 LandmarkCultivars Reveals Asymmetric Sub-genome Selection and Strong Founder GenotypeEffects on Wheat Breeding in China. Molecular Plant, 13(12):1733–1751.
He, F., Pasam, R.,Shi, F., Kant, S., Keeble-Gagnere, G., et al. 2019. Exome sequencing highlightsthe role of wild-relative introgression in shaping the adaptive landscape ofthe wheat genome. Nature Genetics, 51(5): 896–904.
Pont, C., Leroy, T.,Seidel, M., Tondelli, A., Duchemin, W., et al. 2019. Tracing the ancestry ofmodern bread wheats. Nature Genetics, 51(5): 905–911.
Sato, K., Abe, F.,Mascher, M., Haberer, G., Gundlach, H., et al. 2021. Chromosome-scale genomeassembly of the transformation-amenable common wheat cultivar ’Fielder. DNAResearch, 28(3): 1–7.
Walkowiak, S., Gao,L., Monat, C., Haberer, G., Kassa, M. T., et al. 2020. Multiple wheat genomesreveal global variation in modern breeding. Nature, 588(7837):277–283.
Zhu, T., Wang, L.,Rimbert, H., Rodriguez, J. C., Deal, K. R., et al. 2021. Optical maps refinethe bread wheat Triticum aestivum cv. Chinese Spring genome assembly. PlantJournal, 107(1): 303–314.