責編 | 兮
準確的判斷人類基因序列變異的致病性可以使人類基因組序列在精準醫學領域中的潛力得到充分的發揮。儘管全基因組關聯研究(GWAS)探測與疾病相關的罕見錯義變異的能力有限,罕見錯義變化在人類基因組中的地位舉足輕重:超過 99% 的觀察到的人類錯義變異是罕見的(全球次要等位基因頻率或 MAF 低於 0.5%),其中90% 極為罕見(MAF < 10-6)。識別具有破壞性的罕見錯義變異是解讀個人基因組的一個重要和艱鉅的挑戰。
現如今透過高通量的深度突變掃描實驗評估所有可能的錯義變異對單個目標蛋白質功能的影響已經成為可能。但是,在大約4,000 種和人類疾病相關的蛋白質中現僅有不到 1% 有相關的實驗結果,而且實驗往往是在模式生物細胞中進行的。我們可能還需要很長的時間才能獲取完整的高質量的人體細胞深度突變掃描實驗資料。相比之下,利用計算預測模型推斷變異致病性已經可以應用於所有的人類基因。然而,至今所有的計算預測模型效能都尚未能達到臨床應用的標準。
2021年9月21日,加拿大多倫多大學Frederick Roth 團隊,吳穎洲(Yingzhou Wu) 作為第一作者,在The American Journal of Human Genetics期刊上 發表了題為Improved pathogenicity prediction for rare human missense variants 的文章,旨在提升針對罕見人類基因錯義變異致病性的預測模型的效能。在所有同類模型 (比如:Polyphen2, SIFT, REVEL等等)的預測查準率都調整到90%的情況下,文章中新研發的VARITY_R(針對MAF<0.5%的變異) 和 VARITY_ER (針對 MAF<10-6的變異)監督式機器學習模型,比同類模型檢測到至少10%以上的致病變體。
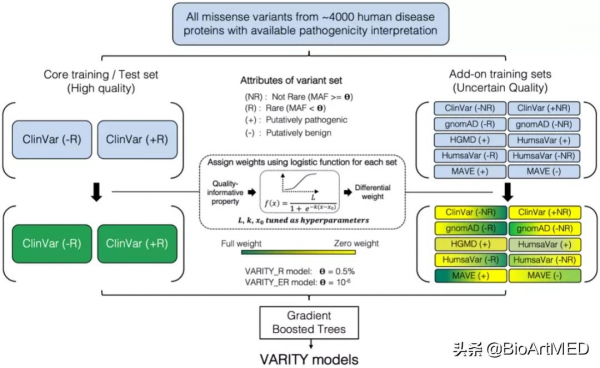
VARITY_R 和VARITY_ER模型的效能提升主要來源於VARITY 機器學習構架(https://github.com/joewuca/varity) 的開發。在監督式學習模型中,當面臨大量的訓練資料但只有一小部分是高質量資料的情況下,VARITY構架可以透過給不同的訓練資料分配與資料質量相關的最優權重來平衡訓練資料質量和數量,從而提升模型的效能。VARITY 構架首先將所有的訓練資料劃分成核心資料(高質量)和附加資料(質量不確定),然後根據需要將核心/附加資料進一步劃分成多個核心/附加資料子集,以便可以對每一個數據子集進行獨立的調整來對模型做出貢獻。對於每一個數據子集,VARITY構架首先透過“移動視窗分析”的方法確立一個或多個“資料質量相關因素”變數,然後針對每個變數設定一個Logistic函式並將該變數作為輸入值, Logistic函式中的引數將作為“權重”超引數和其他演算法相關的超引數一起進行以最佳化模型在核心資料集上的效能為目標的超引數調優。Logistic函式的輸出值將為當前資料子集中的每一條資料分配權重,而最終的模型將在所有已分配權重的訓練資料集上建立。
在構建預測人類罕見基因錯義變異的VARITY_R模型過程中,研究人員採用了VARITY構架,透過不同的資料庫收集到大量的致病性已知的人類錯義變體資料集作為訓練資料 ,但除了從ClinVar 資料庫(有嚴格的稽核流程)採集的資料質量是可以保證的,對提供致病性註釋的其他資源 (比如HumsaVAR,HGMD,gnomAD, MaveDB等等)的準確性不太確定。另外,訓練資料的質量也會被模型目標資料“代表性”所影響。比如這裡旨在建立專門針對人類罕見基因錯義變異的模型,這樣人類常見基因錯義變異也許並不能“代表”人類罕見基因錯義變異,其資料質量也就是對於提高模型效能的作用也就不確定了。因此,研究人員僅僅將ClinVar資料庫中採集到的人類罕見基因錯義變異作為核心資料,而將其他資料庫採集到的資料以及ClinVAR資料庫中的人類常見基因錯義變異作為附加資料。研究人員進一步將核心/附加資料根據資料採集的來源,資料註釋(是否致病),以及變異是否罕見劃分成若干核心/附加資料子集。每一個子集都確定了資料質量相關因素,比如gnomAD資料庫中採集的訓練資料子集研究人員使用了“攜帶同基因合子的人數”作為資料質量相關因素,而從ClinVAR資料庫中採集的訓練資料子集使用了“驗查星數”作為資料質量相關因素。透過超引數調優研究人員確定了和每一個數據質量相關因素相對應的Logistic函式的引數,併為每一個訓練資料子集中的資料賦予了最優的權重,繼而建立的VARITY_R模型。研究人員同時建立了類似的專門針對人類極其罕見的錯義變異的VARITY_ER模型,和VARITY_R的唯一不同就是VARITY_ER僅僅將從ClinVar資料庫中採集到極其罕見的人類錯義變異資料作為核心資料。
VARITY_R和VARITY_ER模型使用的資料特徵包含了進化、蛋白結構以及蛋白質互動作用相關的一系列特徵,使用的機器學習演算法是梯度提升決策樹並採用貝葉斯最佳化的超引數調優方法。在和20種以上的同類模型在和神經發育疾病相關的新生突變資料集,深度突變掃描實驗資料集,以及ClinVar核心資料集(使用巢狀交叉驗證)上做的效能對比中,VARITY_R或者是VARITY_ER取得了具有統計顯著性的優勢。在所有同類模型的預測查準率都調整到90%的情況下,VARITY_R和 VARITY_ER模型比同類模型檢測到至少10%以上的致病變體。
研究人員使用VARITY_R和VARITY_ER模型對將近18,000種人類蛋白質可能出現的所有基因錯義變異的致病性做了預測,並對每一個變異的預測結果做了夏普利值分析,提供了每一個變異的預測結果中每個特徵的貢獻。所以的預測結果可以透過varity.varianteffect.org查詢並下載。
本文來自BioArtMED微信公眾號,更多生物領域前沿資訊等你來發現!
轉載須知
【原創文章】BioArtMED原創文章,歡迎個人轉發分享,未經允許禁止轉載,所刊登的所有作品的著作權均為BioArtMED所擁有。BioArtMED保留所有法定權利,違者必究。