▲第一作者:Matthew J. Mitcheltree, Amarnath Pisipati, Egor A. Syroegin
通訊作者:Andrew G. Myers, Yury S. Polikanov
通訊單位:Harvard University, University of Illinois at Chicago
DOI: 10.1038/s41586-021-04045-6
背景介紹
缺乏針對耐抗生素細菌的有效新藥是一個日益引起全球公共衛生關注的問題。50多年來,對新抗生素的研究一直嚴重依賴於對天然產物的化學修飾(半合成),這種方法不足以應對迅速演變的耐藥性威脅。半合成修飾在多功能抗生素中通常範圍有限,通常增加分子量,很少允許底層支架的修飾。而如果設計得當,全合成可以很容易地解決這些缺點。
本文亮點
● 本文報道了結構導向設計和基於元件的剛性氧雜嘌呤支架合成,當連線到克林黴素的氨基半乳糖殘基時,產生一種特殊的效能和光譜活性的抗生素,作者將其命名為iboxamycin。
● Iboxamycin對ESKAPE病原體有效,包括表達Erm和Cfr核糖體RNA甲基轉移酶的菌株,以及對所有臨床相關針對大核糖體亞基的抗生素(即大環內酯類、林可醯胺類、酚類、惡唑烷酮類、胸膜多菌素類和鏈黴素)產生耐藥性的基因產物。
● 作者對伊博黴素與天然細菌核糖體以及Erm-甲基化核糖體複合物的X射線晶體學研究,揭示了這種增強活性的結構基礎,包括在抗生素結合上的m6 2A2058核苷酸的置換。
● Iboxamycin是口服生物可利用的,對治療小鼠的革蘭氏陽性和革蘭氏陰性細菌感染都是安全有效的,本文證明了化學合成在耐藥性不斷增加的時代提供新抗生素的能力。
圖文解析
核糖體是細菌中的一個主要抗生素靶點,林可醯胺類是現代藥典證明必不可少的幾個核糖體靶點類別之一。林可黴素(1;圖1a)是該類的創始成員,於1963年首次從內布拉斯加州土壤鏈黴菌中分離出來,並很快被發現用於治療鏈球菌、肺炎球菌和葡萄球菌感染。在1的早期半合成修飾中,氨基糖殘基C7位置的立體轉化脫氧氯化產生了現在稱為克林黴素的抗生素(2;圖1a),這是一種具有改進的藥代動力學性質和增強的活性譜的分子,已在很大程度上取代人類藥物中的林可黴素。自美國食品和藥物管理局於1970年批准克林黴素以來,已經探索了林可醯胺發現的半合成和全合成方法,產生了含有六元和七元氨基醯基殘基的候選藥物,每種藥物都具有擴充套件的活性譜(例如,3;圖1a)。在大環內酯類抗生素的合成中,一個關鍵片段偶聯的立體選擇性醛醇反應啟發並使這些探索成為可能。因此,(R,R)-4烯醇化,手性甘氨酸等價物,然後加入雙親電試劑,導致依次的順式-醛醇加成,然後自發的分子內N-烷基化,生成β-羥基脯氨酸衍生物6(產率64%,2.3 g)(圖1c)。
▲圖1、一種新型抗生素支架的演變。
肉質培養藥敏試驗顯示,IBX對廣泛耐藥菌株具有活性,包括ESKAPE病原體和對林可胺類抗生素耐藥的erm-、cfr-和lsaA-表達菌株(圖2a)。腸球菌病原體可導致廣泛且危及生命的醫療相關感染,且對林可醯胺類藥物不敏感,也廣泛易感IBX:在糞腸球菌中,IBX克服了靶保護基因lsaA介導的固有耐藥性(圖2a),針對一組對萬古黴素、利奈唑胺、強力黴素、阿奇黴素和左氧氟沙星具有不同耐藥性的糞腸球菌和糞腸球菌菌株,MICs≤2 μg mL−1(圖2b)。耐碳青黴烯類大腸桿菌、肺炎克雷伯菌和鮑曼不動桿菌等ESKAPE高優先順序病原體,包括表達廣譜β-內醯胺酶或氨基糖苷甲基化酶(arm)基因的菌株,對ESKAPE大多敏感(MIC≤16 μg ml−1;圖2c)。在一個例子中,IBX對一株耐頭孢菌素、氟喹諾酮、四環素和氨基糖苷類抗生素的大腸桿菌表現出活性(MIC = 8 μg ml−1;圖2c)。接下來,作者研究了IBX在小鼠中性粒細胞減少性大腿感染模型中的療效,使用標準菌株化膿性鏈球菌和鮑曼鏈球菌,臨床菌株耐甲氧西林金黃色葡萄球菌,臨床菌株大腸桿菌顯示氨基糖苷、四環素,氟喹諾酮和第三代頭孢菌素多重耐藥。在每個實驗中,當與載體相比時,IBX在治療後12小時內實現了細菌負荷的統計顯著減少(圖2d)。最後,在一個全身性感染模型中,作者研究了IBX從腹腔注射化膿性鏈球菌ATCC 19606致死攻擊中拯救小鼠的能力。在該實驗中,IBX在所有劑量水平下耐受性良好,並挽救了受感染的小鼠,所有接受3或10 mg kg−1 IBX治療的小鼠都存活下來(圖2e)。
▲圖2、廣譜抗生素IBX的體外和體內抑菌活性。
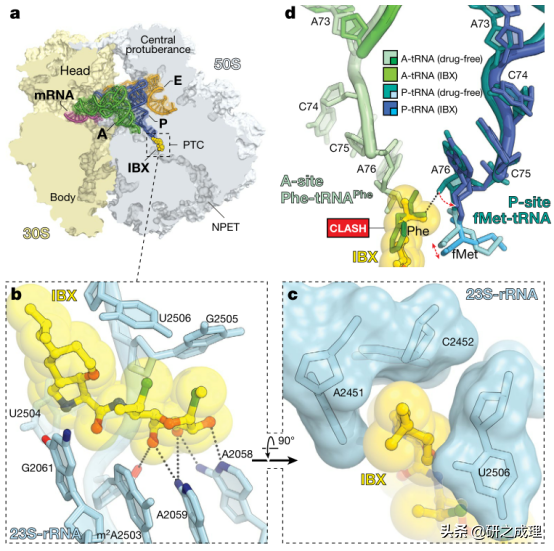
為了闡明活性提高的結構基礎,作者使用X射線晶體學確定了與細菌核糖體結合的IBX的結構。無偏的FO−FC差值傅立葉圖顯示了與IBX特徵化學特徵相似的正電子密度峰,並證實抗生素結合在大的核糖體亞單位內的正結合口袋中(圖3A),跨越肽基轉移酶中心(PTC)並延伸到新生肽出口隧道(NPET)。與林可黴素和林可黴素一樣,一個氫鍵網路將IBX的氨基半乳糖固定在NPET核苷酸A2058、A2059和A2503上(圖3b)。類似地,IBX的陽離子氨基醯殘基佔據了由PTC殘基G2061和U2504形成的親水口袋(圖3b),取代了在無藥核糖體中觀察到的二價鎂離子。與假設一致,核糖體結合的IBX的氧烷環與克林黴素的正丙基原子緊密重疊,並呈現7'-異丁基與23S rRNA殘基A2451和C2452形成的A位裂口相互作用(圖3c)。值得注意的是,IBX的7'-異丁基取代基在A位裂縫中延伸足夠深,不僅與進入的氨基酸重疊(如克林黴素的正丙基,儘管程度較輕),而且與p位氨基酸重疊(圖3d)。
▲圖3、IBX與70S核糖體、mRNA和tRNAs複合體的結構。
IBX對含有erm抗性基因的細菌的活性促使作者確定了與含有m6 2A2058的核糖體結合的IBX的晶體結構。值得注意的是,相應的電子密度圖顯示,IBX與甲基化核糖體的結合方式幾乎與其在野生型核糖體中的位置相同,而m6 2A2058相對於其典型位置經歷了約2Å的移動,以適應抗生素(圖4)。A2058-N二甲基化以及之前未知的置換,破壞了殘基和林可醯胺的氨基乙醯氨基部分之間通常形成的氫鍵(圖4d);這種破壞似乎足以破壞克林黴素與Erm修飾核糖體的結合。
▲圖4、Iboxamycin(IBX)與Erm甲基化的70S核糖體結合的結構。
原文連結:
https://www.nature.com/articles/s41586-021-04045-6