作者 | 陳彩嫻
近日,就讀於世界計算機專業頂級院校卡內基梅隆大學(CMU)的 1 名中國博士生開發了一個機器學習演算法 Higashi,可以詮釋人體細胞核中基因組的摺疊方式、以及這些摺疊如何影響基因的表達,研究登上了《Nature Biotechnology》!

透過對蛋白質與DNA束的摺疊與排序瞭解,專家可以瞭解哪些基因得到表達、哪些基因又遭到了抑制,從而判斷人體的健康狀況。但此前,科學家對這個過程並不瞭解。
而 1 名 CMU 在讀的中國博士生,用一種基於超圖表示學習(hypergraph representation learning)的演算法破解了這一謎題!超圖表示學習是一種機器學習方法,原先主要用在 app 音樂推薦或 3D 物體識別。
這名博士生的名字為張若弛,本科畢業於清華大學,目前就讀於 CMU 的計算生物學系(CBD),追求“幽默科研”,在這次研究中還將演算法命名為“Higashi”(一種日本甜食)。Ruochi Zhang的博士導師為 CMU 知名華人學者馬堅,另一位合作者也是中國學生,叫 Tianming Zhou(本科畢業於清華大學)。
該研究專案由美國國立衛生研究院(NIH)資助。2020年,馬堅獲得 NIH 的 1000 萬美元科研資助,在 CMU 成立一個多研究中心,旨在瞭解細胞核的 3D 結構、及其結構的變化如何影響細胞在健康與疾病中的功能。

圖注:從左到右,Ruochi Zhang、Tianming Zhou與馬堅
1 Higashi 演算法如何工作?
染色體由稱為“染色質”的DNA-RNA-蛋白質複合物組成。該複合物會摺疊並自行排列來適應細胞核。在這個過程中,每一種成分的功能元素都會更緊密地結合在一起,從而影響了基因的表達方式,啟用或抑制特定的遺傳特徵。
基因組組織的可變性對基因表達與細胞狀態的影響很大。
Higashi 演算法與新興技術(單細胞 Hi-C)配合使用,可以建立單細胞中染色質相互作用的快照。Higashi 對複雜組織和生物過程中單個細胞的染色質組織進行了更詳細的分析,同時深究了染色質的相互作用如何因細胞而異。這些分析使科學家能夠看到細胞與細胞之間染色質摺疊和組織的詳細變化——包括那些可能很微妙但對健康有影響的變化。
Higashi 的關鍵演算法設計是將 scHi-C 資料轉換為超圖(如圖 1a)。在轉換的過程中,超圖會保留來自 scHi-C 接觸圖的單細胞解析度和 3D 基因組特徵。
據 CMU News 報道,Higashi演算法是第一項在超圖上使用複雜神經網路來對單細胞基因組組織進行高畫質分析的技術。普通的圖只是將兩個頂點連線到一個交叉點(即“邊”,edge),而超圖是將多個頂點連線到邊。
詳細來說,嵌入 scHi-C 資料的過程就相當於學習超圖的節點嵌入,而輸入 scHi-C 接觸圖就變成了預測超圖中缺失的超邊。
在 Higashi 中,他們使用了最新開發的 Hyper-SAGNN 架構。這是一個通用的超圖表示學習框架,專門針對 scHi-C 分析進行了大量的新開發。
Higashi 有 5 個主要組成部分:
1)他們將 scHi-C 資料集表示為超圖,其中,每個細胞和每個基因組 bin 分別表示為細胞節點和基因組 bin 節點。單細胞接觸圖中的每個非零條目都被建模為連線相應細胞和該特定染色質相互作用的兩個基因組位點的超邊(圖 1a)。這種形式集合了 scHi-C 的嵌入和資料插補。
2)他們基於構建的超圖訓練超圖神經網路 (NN)。
3)將訓練好的超圖神經網路中提取單元節點的嵌入向量用於下游分析。
4)使用經過訓練的超圖 NN 來插補單細胞 Hi-C 接觸圖,並結合細胞之間的潛在相關性來增強整體插補,從而更詳細地表徵 3D 基因組特徵。
5)通過幾種新的計算策略,比較了跨單個細胞的 A/B 區室分數和 TAD 樣域邊界,以促進對這些大規模 3D 基因組特徵的細胞間變異性及其對基因轉錄的影響的分析。
2 識別 3D 基因組結構
團隊試圖用 Higashi 估算的接觸圖來識別細胞型別特定的 3D 基因組結構。
單細胞 Hi-C (scHi-C) 方法可以識別 3D 染色質組織的細胞間變異性,但分析已測量的染色質互動的稀疏性具有一定挑戰。馬堅團隊所提出的 Higashi 演算法可以結合單個細胞之間的潛在相關性,增強接觸圖的整體插補。
他們的分析表明,基於 Higashi 推算接觸圖計算的單細胞絕緣分數具有分離複雜細胞型別的能力,而基於原始接觸圖的單細胞絕緣評分不能有效區分細胞型別。

圖 2:Higashi 能夠以單細胞解析度詳細描述 3D 基因組特徵及其與基因轉錄的聯絡。
Higashi 可以識別單個細胞中的多尺度 3D 基因組特徵,從而精確地描繪細胞間的變異性。在關於人類前額葉皮層的 scHi-C 資料集中,Higashi 可以確定 3D 基因組特徵與細胞型別特異性基因調控之間的聯絡。
他們還發現 SULF1 是區分 L6 亞型與其餘興奮性神經元亞型(L2/3、L4 和 L5)的標記基因,與周圍細胞型別特異性 TAD 樣域邊界具有很強的相關性。TAD 樣域邊界存在於 93.2% 的 L6 細胞中,但其餘的興奮性神經元亞型中僅有 65.3%。這些結果為人類前額葉皮層細胞型別的標記基因調控以及 3D 基因組結構和功能之間的聯絡提供了新的見解。
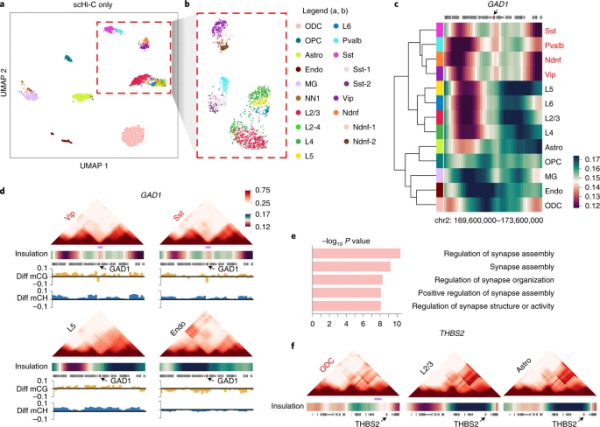
圖 3:Higashi 使用來自人類前額葉皮層的 scHi-C 資料識別複雜的細胞型別和細胞型別特異性 TAD 樣域邊界。
最後,他們希望知道 Higashi 確定的細胞型別特異性 TAD 樣域邊界附近的基因是否具有不同的功能作用。結果證明,Higashi 在使用 scHi-C 資料有效識別複雜組織中的細胞型別和細胞型別特異性 3D 基因組特徵方面具有獨特優勢。
這個分析表明,Higashi 在揭示細胞型別特異性 TAD 樣域邊界方面有強大潛力,極大地促進了 3D 基因組結構在調節細胞型別特異性基因功能中的作用分析。
此外,據 Nature 報道,Higashi 演算法還可以用於分析單細胞多路染色質相互作用與其他多模式單細胞組學資料。
3 總結
馬堅團隊開發的機器學習演算法 Higashi 在嵌入與插補方面有極大優勢。
透過 scHi-C 接觸圖的資料增強改進,他們在 Higashi 演算法中開發了能系統分析可變多尺度 3D 基因組特徵(A/B 區室分數和 TAD 樣域邊界)的方法,並證明了這些特徵對基因轉錄的影響。
透過應用來自人類前額葉皮層的 scHi-C 資料集,Higashi 能夠識別複雜的細胞型別,並揭示與細胞型別特異性基因調控有密切聯絡的細胞型別特異性 TAD 樣域邊界。
Higashi 的關鍵演算法創新是將 scHi-C 資料轉化為超圖,與現有方法相比具有獨特的優勢:
首先,這種轉換保留了 scHi-C 的單細胞精度和 3D 基因組特徵。
其次,將整個 scHi-C 資料集建模為超圖,而不是將每個接觸圖建模為單獨的圖,允許跨細胞協調資訊,透過利用細胞之間的潛在相關性來改善嵌入和插補。
最後,雖然他們主要關注 scHi-C 資料,但 Higashi 中的超圖表示高度可以推廣到其他單細胞資料型別。
不過,他們指出,作為一種資料驅動演算法,Higashi 也有不少需要改進的地方。比如,它至少需要一箇中等大小的 scHi-C 資料集來實現高效能。此外,由於單細胞 3D 基因組結構性質的高度多樣化,Higashi在遠端互動的估算方面仍有很大的改進空間。
但他們也談到,為了在單細胞解析度下更全面地描繪 3D 基因組組織,Higashi 可以擴充套件到分析高階染色質結構的單細胞分析——例如,最近開發的 scSPRITE34 探測多路染色質相互作用。
參考連結:
1.https://www.cs.cmu.edu/news/2021/higashi-algorithm
2.https://www.nature.com/articles/s41587-021-01034-y
3.https://www.cmu.edu/news/stories/archives/2020/october/new-center-cell-nucleus.html