人體發育過程中形成的成百上千種細胞型別有著近乎一樣的遺傳資訊(DNA),但卻能演化出全然不同的細胞外形和功能。表觀遺傳機制(Epigenetics)調控的基因轉錄參與決定了每個細胞型別的特異基因表達譜【1,2】,並進而決定其形態及功能。在眾多的表觀遺傳機制中,兩類蛋白複合體PRC1 和PRC2參與了兼性異染色質(facultative heterochromatin)的建立和維持,這一過程在發育過程中至關重要【3,4】。長久以來,研究者們認為PRC1 和PRC2透過調節組蛋白修飾和染色質結構,主要參與維持基因轉錄的抑制狀態。近年來越來越多的證據表明一類非經典PRC1(ncPRC1)出乎意料地參與基因轉錄的啟用【5-7】,但具體的作用機制以及在發育過程中的作用還有待進一步研究。
2021年10月11日,紐約大學醫學院/霍華德休斯醫學研究所Danny Reinberg實驗室(第一作者為柳三雄)在雜誌以長文形式線上發表了一篇題為“”的研究論文。該研究詳細闡述了非經典PRC1(ncPRC1)啟用基因轉錄的分子機制。
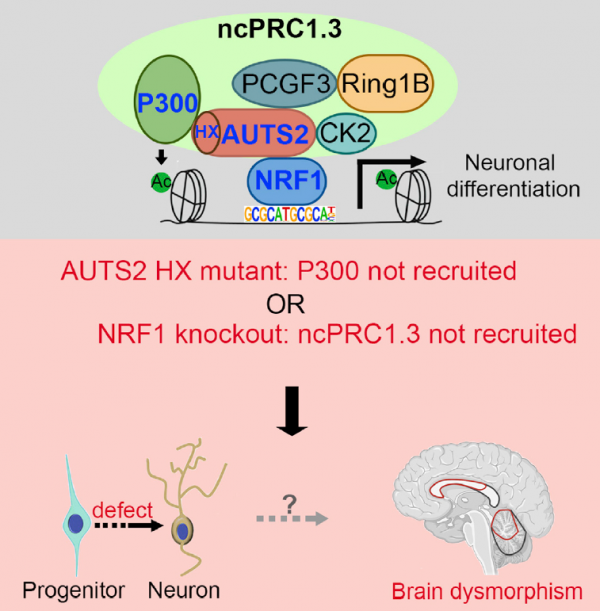
Danny Reinberg實驗室之前首次報道自閉症相關因子AUTS2是非經典PRC1(ncPRC1.3/1.5)的重要組成部分,正是由於AUTS2結合酪蛋白激酶CK2和轉錄共啟用因子P300才使得非經典PRC1具備啟用轉錄的特性【5】。本研究致力於更深入探討這一機制。巧合的是,作者發現七位帶有AUTS2突變的病例,他們的表型與AUTS2完全缺失所造成的病例表型大有不同,其中五位病人帶有的AUTS2突變(造成AUTS2蛋白的單點突變或8個氨基酸缺失)位於其蛋白的一個小的功能域(HX repeat domain)[圖1]。更重要的是這些病人在經外顯子測序確定突變位點之前,被臨床診斷為Rubinstein-Taybi syndrome(RSTS),一種主要由P300/CBP突變或缺失造成的多器官發育障礙【8】。考慮到之前報道的AUTS2和P300之間的相互作用,作者推定HX repeat domain是介導這一相互作用的關鍵部位。透過在內源AUTS2基因上引入相關突變以及一系列分子生化實驗,作者證實HX repeat domain介導了AUTS2和P300的相互作用,以及由這一相互作用所引起的基因轉錄啟用。
由於已發現的非經典PRC1(ncPRC1.3/1.5)的組成蛋白中並不含有能結合DNA的功能域,作者進一步探索了該複合物如何特異性地結合靶標基因。首先,針對AUTS2-PRC1結合的DNA位點的motif分析,發現轉錄因子NRF1結合DNA的motif顯著富集。其次,大約75%的AUTS2結合的DNA位點和NRF1 結合位點重疊。最後, 透過比較AUTS2 在對照細胞和NRF1敲除細胞中的ChIP-Seq訊號,發現AUTS2在其絕大多數目標位點的結合顯著降低或消失。這些證據充分表明轉錄因子NRF1介導了AUTS2-PRC1在染色質上的定位。值得一提的是,NRF1條件性敲除的小鼠也表現出與RSTS類似的神經系統發育障礙。
最後作者初步探索了AUTS2的突變是如何引起疾病發生的。透過構建AUTS2敲除,內源性AUTS2基因突變或NRF1敲除的mESC,並進一步誘導神經元分化,結合最先進的單細胞測序技術(scRNA-Seq, smart-seq3)比較了最終分化形成的不同細胞型別的組成以及基因表達差異。這些實驗表明上文所揭示的分子機制對於progenitor到neuron分化過程中所需要的基因轉錄啟用至關重要,併為理解AUTS2 HX repeat domain的突變如何引起神經發育障礙提供了重要線索(下圖)。
(該研究結論的示意圖)
綜上所述,該研究詳細闡述了轉錄因子NRF1特異性地招募AUTS2-PRC1在染色質上的定位,並透過HX repeat domain所介導的AUTS2-P300相互作用決定了靶標基因的轉錄啟用,這一過程對於大腦的正常發育至關重要。
該論文通訊作者為紐約大學醫學院/霍華德休斯醫學研究所研究員Danny Reinberg,第一作者為實驗室博士後柳三雄。該研究得到西雅圖兒童醫院William B. Dobyns實驗室,德克薩斯大學健康科學中心Chai-An Mao實驗室的大力幫助。
原文連結:
https://doi.org/10.1016/j.molcel.2021.09.020
參考文獻
1.Li, E. Chromatin modification and epigenetic reprogramming in mammalian development. 3, 662–673 (2002).
2.Perino, M. & Veenstra, G. J. C. Chromatin Control of Developmental Dynamics and Plasticity.38, 610–620 (2016).
3.Trojer, P. & Reinberg, D. Facultative Heterochromatin: Is There a Distinctive Molecular Signature? 28, 1–13 (2007).
4.Schuettengruber, B., Bourbon, H.-M., Di Croce, L. & Cavalli, G. Genome Regulation by Polycomb and Trithorax: 70 Years and Counting. 171, 34–57 (2017).
5.Gao, Z. et al. An AUTS2–Polycomb complex activates gene expression in the CNS. 516, 349–354 (2014).
6.Cohen, I. et al. PRC1 Fine-tunes Gene Repression and Activation to Safeguard Skin Development and Stem Cell Specification. 22, 726-739.e7 (2018).
7.Loubiere, V., Papadopoulos, G. L., Szabo, Q., Martinez, A.-M. & Cavalli, G. Widespread activation of developmental gene expression characterized by PRC1-dependent chromatin looping. 6, eaax4001 (2020).
8.Stevens, C. A. Rubinstein-Taybi Syndrome. in GeneReviews (eds. Adam, M. P. et al.) (University of Washington, Seattle, 1993).
來源: BioArt