近年來,單細胞測序的快速發展極大推進了我們對生物系統的認知。這項技術的巨大潛力促使計算生物學家開發了一系列的分析工具,但大部分分析工具要求程式設計背景。今天,小編為大家介紹一款免費且幾乎不需要程式設計背景,即可實現單細胞分析的工具,cellxgene。
Cellxgene有兩個產品,cellxgene data portal用於探索已發表的資料;cellxgene desktop用於探索個人單細胞資料。小編主要對cellxgene desktop進行描述,並在接下來的內容中簡稱為cellxgene。
單細胞分析過程中,包含測序結果的fastq檔案轉化為細胞-基因表達量矩陣、質控(Quality Control)結束後即可進行聚類(Clustering)。聚類將表達模式相近的細胞歸為一類,並給每個細胞分群分配一個數字ID(Fig. 1a)。為每個由數字代表的細胞群打上一個代表細胞身份的標籤的過程稱為細胞型別註釋(Fig. 1b),該過程有助於從生物學角度理解聚類結果。Cellxgene即可實現為細胞打標籤的過程。此外,cellxgene還可實現成分分析,差異分析等,本篇小指南主要從細胞型別註釋和成分分析兩方面介紹。
個人資料
如果你的資料是h5ad格式並滿足一定要求,則該資料可直接被cellxgene使用。
h5ad檔案
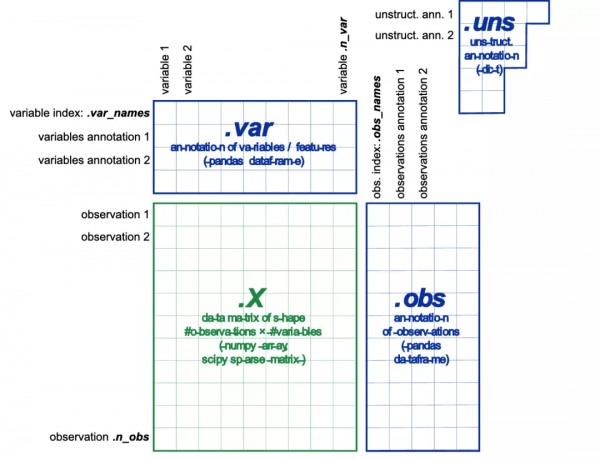
h5ad檔案提供了一種可擴充套件的方式來記錄資料及其註釋 (annotation)。單細胞測序的h5ad被分割成X, obs, var, uns等多個部分 (Fig. 2),分別儲存不同的資訊。其中X是表達量矩陣;obs包含細胞元資料,即從不同方向描述細胞的結果,例如細胞所屬tissue;var包含基因元資料,即從不同方向描述基因的結果,X, obs, var均以dataframe格式儲存;uns是非結構化註釋,更多解釋可查詢h5ad(AnnData)。
h5ad檔案需滿足的要求
1. X是矩陣資料(通常是原始或經過normalization的資料)
2. 至少一種降維方式(例如 tSNE, UMAP)的結果
3. 每個細胞需要一個唯一的識別符號
4. 每個基因需要一個唯一的識別符號
官方資料
除對自己的資料進行註釋、探索外,cellxgene data portal也儲存了許多公共資料,本篇小指南使用肺部資料對該軟體的各種功能進行解釋(Fig.3),由於需要展示細胞型別註釋過程,已刪除原註釋。
Cellxgene需要python 3.6以上的版本及最新的Google Chrome瀏覽器。Cellxgene無桌面版,只能開啟終端,輸入以下命令安裝並啟動cellxgene,
# 安裝
pip install cellxgene
# 啟動
cellxgene launch h5ad_path/mydataset.h5ad --openh5ad_path代表存放h5ad檔案的路徑,可右鍵h5ad檔案️ 屬性️ 位置,位置給出的路徑即為存放h5ad檔案的路徑,實際啟動時,使用該路徑取代上述命令中的h5ad_path即可。
Cellxgene提供了一個強大且清晰的介面供使用者探索自己的單細胞資料,該章節從多種目的出發,解釋cellxgene的使用。
1. Cellxgene介面介紹
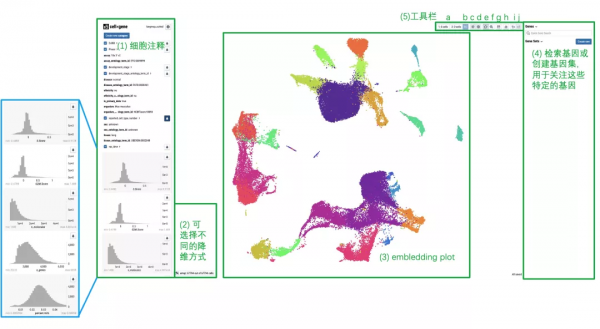
啟動cellxgene後,chrome瀏覽器會自動開啟Fig. 4中的介面,該介面可被大致分為五部分,
(1) 細胞元資料
h5ad檔案中obs部分(即對細胞的描述)均在左側面板展現出來,左側面板上下兩部分分別透過分類和數值統計描述細胞。例如reported_cell_type_number為利用聚類對細胞分群后,為細胞分配的數字ID;n_genes表示細胞表達的基因數量分佈。
右側水滴狀圖標表示可根據該分類對細胞染色。點選每種分類的下拉鍵頭,可檢視該分類下所有型別、該型別對應的細胞數、用以代表該型別的顏色。點選數值型統計右側的染色鍵,同樣可以根據對應引數對細胞染色。
(2) 降維方式:cellxgene將基因的表達量資料降維後展示在二維embledding plot上,如h5ad檔案包含多種降維方式的結果,則多種降維方式可選擇,該資料集只包含umap;
(3) embledding plot: 降維後的圖,每個點代表一個細胞,相似的細胞聚集在一起;
(4) 基因:檢索感興趣的基因,檢視該基因的表達情況;
(5) 工具欄:實現細胞選擇,計算marker基因等功能,具體為:
a) 選擇進行差異分析的細胞集1和細胞集2;
b) 進行差異分析,並給出差異基因
c) 選擇子細胞集
d) 還原子細胞集為整個資料集
e) 套索 (lasso) 選擇工具
f) 縮放+移動畫布
g) 展示分類標籤。基於不同標準對細胞染色後,該鍵可在embledding plot中為細胞新增標籤
h) 篩選
i) 撤回
j) 重做
2.細胞型別註釋
細胞型別註釋是依據標誌物(Marker)為細胞群打上代表細胞身份的標籤的過程。標誌物是指在已知細胞型別中特異性表達的基因。理想情況下,每個細胞群將獨特地表達一個細胞型別的標誌物。標誌物和細胞型別之間的對應關係可從文獻或資料庫中獲得。
利用cellxgene為細胞群打標籤可透過以下步驟完成:
(1)建立新分類方式,cell_type,用於儲存細胞型別註釋的結果(Fig.5)。
a. 點選create new category建立新分類
b. 複製reported_cell_type_number,建立新分類cell_type
(2)為細胞群打標籤(Fig.6)
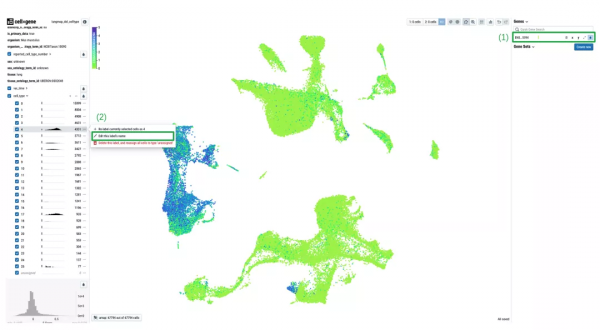
a.檢視來源於文獻或資料庫的marker的表達情況。該圖中以ENSMUSG00000045394(即Epcam)為例,Epcam是肺部上皮細胞(Epithelial cell)的marker,該marker在細胞分群 4,6,7,17中分佈較廣。
b.將細胞分群 4,6,7,17修改為Epithelial cell。以此類推,尋找更多marker,並將所有細胞分群逐一修改。利用其他細胞型別對應的marker逐一註釋過程中,可能會與前期註釋結果產生矛盾,在這種情況下,需要尋找更多證據對細胞進行最終註釋。
3. 成分分析
細胞型別的比例差異往往具有生物學意義,例如細胞型別構成的差異可以揭示疾病機制、癌症的免疫反應和發育過程。在細胞水平,從組成結構的角度來分析聚類資料的過程即為成分分析。Cellxgene提供一種非常方便的方式進行成分分析。例如檢視不同型別細胞在不同年齡段樣本中的佔比,點選reported_cell_type_number右側的染色鍵,然後點選var_time的下拉鍵頭即可檢視 (Fig. 7)。此外,cellxgene還可分組檢視數值型統計在不同分組中的分佈狀況,如Fig. 8。
以上即為利用cellxgene進行細胞型別註釋和成分分析的過程,差異分析和尋找marker的小指南敬請期待!