近日,南方科技大學生命科學學院副教授翟繼先團隊利用奈米孔單分子RNA測序技術繪製了擬南芥全基因組水平轉錄終止圖譜。相關成果以“Landscape of transcription termination in Arabidopsis revealed by single-molecule nascent RNA sequencing”為題發表在國際知名學術期刊Genome Biology上。
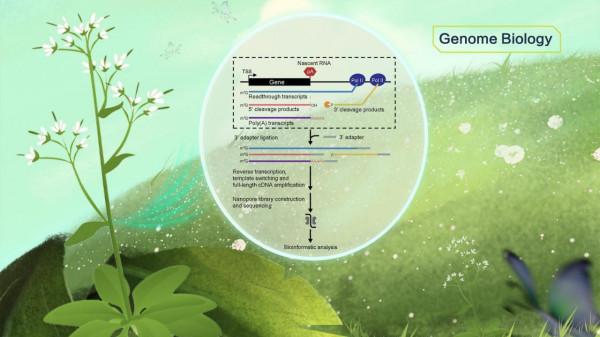
奈米孔單分子RNA測序技術能同時捕獲包括已轉錄過polyA位點的 readthrough轉錄本、完成3’末端切割的5’或3’切割產物、以及已完成或正在進行多腺苷酸化(polyadenylation)的轉錄本在內的多種轉錄終止過程中的RNA中間產物(圖1)。結果顯示,擬南芥不同基因的轉錄終止範圍分佈很廣,從50 nt到超過1000 nt。研究團隊應用此方法檢測atxrn3和fpa,以及DNA甲基轉移酶met1等一系列突變體,發現在atxrn3中轉錄終止距離變長,fpa中有出現多基因嵌合轉錄本, met1中基因區CG甲基化的丟失對轉錄終止沒有明顯影響。
轉錄終止是RNA合成中關鍵的最後一步,在此期間新生RNA從RNA聚合酶II(Pol II)和DNA模板中釋放出來。轉錄終止對於防止轉錄順讀(transcription readthrough)進入下游基因是至關重要的。該領域幾十年的廣泛研究提出了變構模型(allosteric/antiterminator model)、魚雷模型(torpedo model),以及後來結合這兩種模型的統一模型(unified model),以解釋多腺苷酸訊號依賴的終止機制。成熟mRNA的3’端是由新生RNA的末端切割決定的,而不是聚合酶轉錄終止的位置。3’末端切割(3’ cleavage)為核酸外切酶Xrn2提供入口,使其從5’端開始降解與RNA聚合酶II(Pol II)相連的RNA,直至“追趕”上聚合酶本身時引發轉錄終止。儘管已經有一些基於Illumina測序來分析新生RNA的方法,但他們的目的主要捕獲的是在轉錄延伸過程的RNA,而不是轉錄終止過程中產生的RNA中間產物;並且由於測序讀長的限制,這些方法不能很好區分readthrough轉錄本是否完成3’末端切割。

圖1. 單分子新生RNA測序技術FLEP-seq區分轉錄終止RNA中間體示意圖
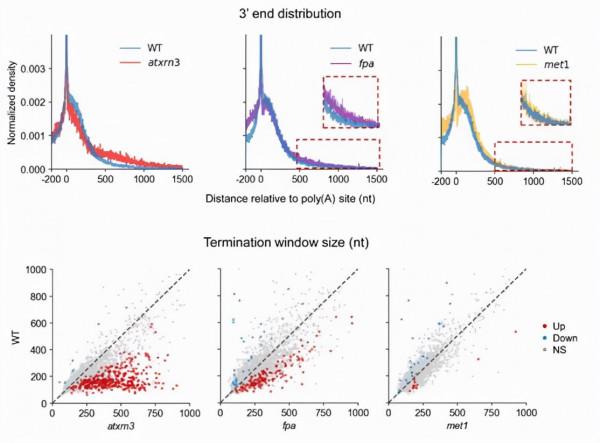
轉錄終止隨機發生在polyA位點下游,從Pol II在polyA位點到其在釋放的一段區間被稱“終止視窗(termination window)”。資料顯示,在擬南芥基因組中各基因的終止視窗大小分佈範圍很廣,從50 nt到超過1000 nt(圖2)。團隊還觀察到下游鄰近的tRNA基因可有效促進上游基因轉錄終止,這表明tRNA基因啟動子區域周圍的染色質結構可能會限制Pol II的延伸(圖2)。

圖2. 擬南芥中轉錄終止視窗大小分佈(左圖)和tRNA有效促進上游基因轉錄終止(右圖)
研究團隊分別構建了與轉錄終止相關基因AtXRN3和FPA,以及與DNA甲基化相關基因Met1的FLEP-seq突變體文庫,發現在atxrn3突變體中轉錄終止發生了延遲,而在fpa突變體中發現了嵌合轉錄本的積累;但在met1突變體中CG甲基化的丟失對轉錄終止沒有明顯影響(圖3)。
此外,團隊還發現在個別位點中上游基因的已經完成3’切割的readthrough轉錄本可以繼續延伸並進入下游基因區域,這些轉錄本可以產生正常剪接和多腺苷酸化的RNA,表明readthrough本身就能驅動下游基因轉錄本的產生,這突出了AtXRN3介導的轉錄終止在擬南芥基因組中的重要性(圖4)。
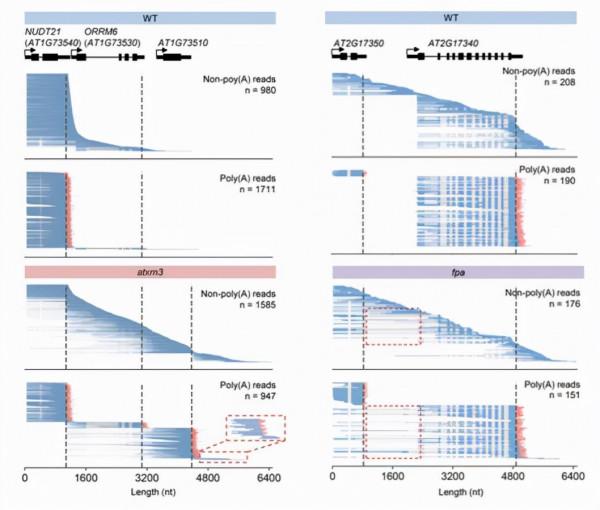
圖4. FLEP-seq檢測atxrn3(左圖)和fpa突變體(右圖)轉錄終止的例子
翟繼先為該論文的通訊作者,南科大生命科學學院博士研究生莫偉鵬和劉波為該論文共同第一作者,中科院遺傳與發育生物學研究所研究員曹曉風、副研究員鄧嫻,南科大生命科學學院講席教授郭紅衛、研究助理教授龍豔萍、賈津布等參與了該項工作。南科大是論文第一單位。該研究得到了科技部國家重點研發計劃、廣東省創新創業團隊、深圳市科創委、廣東省植物細胞工廠分子設計重點實驗室以及深圳市高等院校穩定支援計劃基金的資助。
論文連結:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-021-02543-4
供稿:生命科學學院
通訊員:付文卿
主圖:丘妍
編輯:朱增光